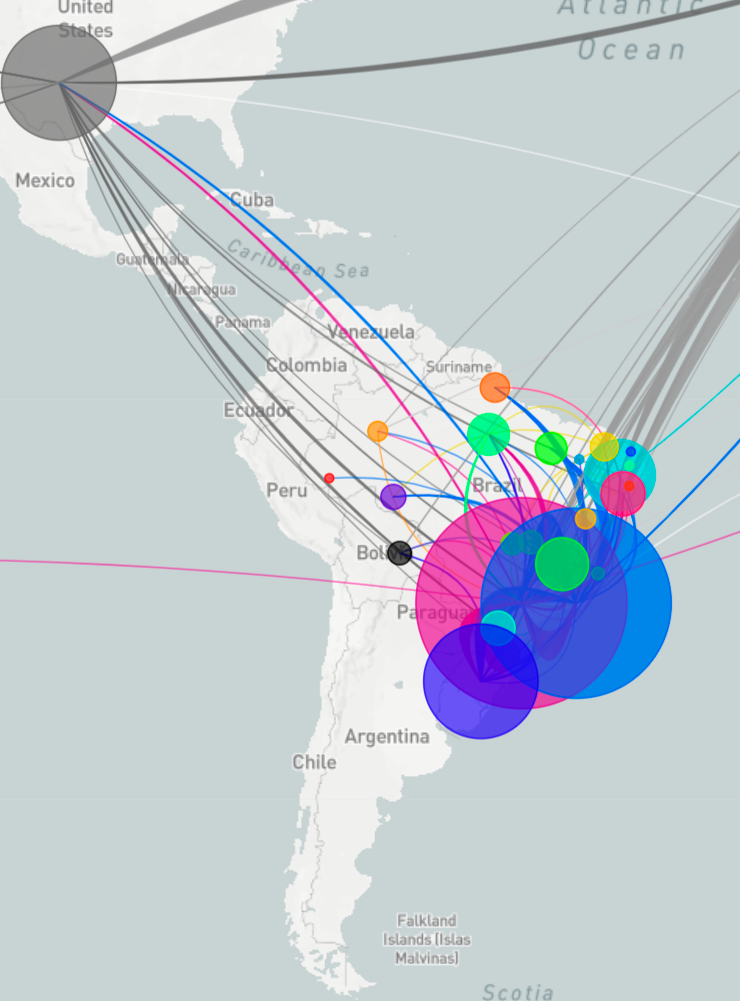
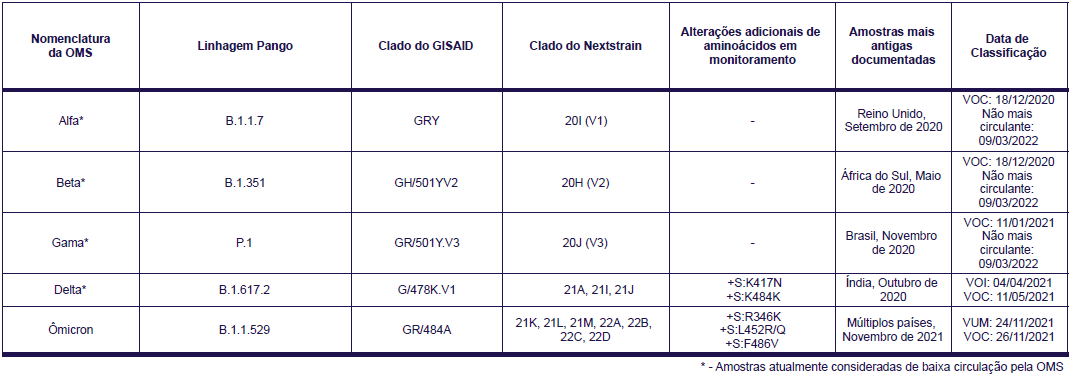
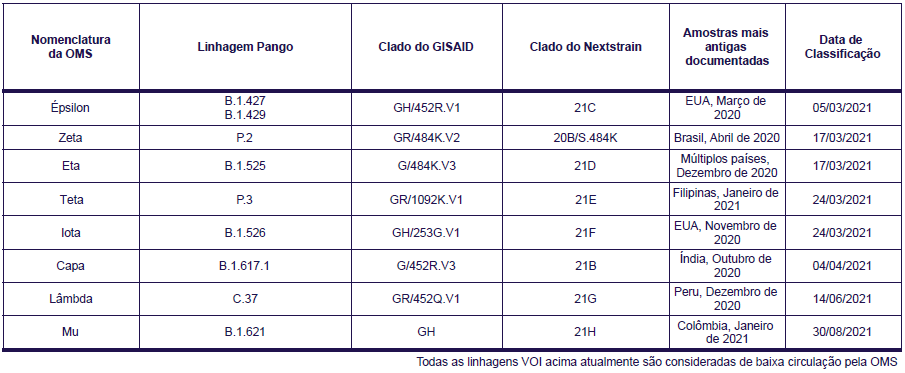
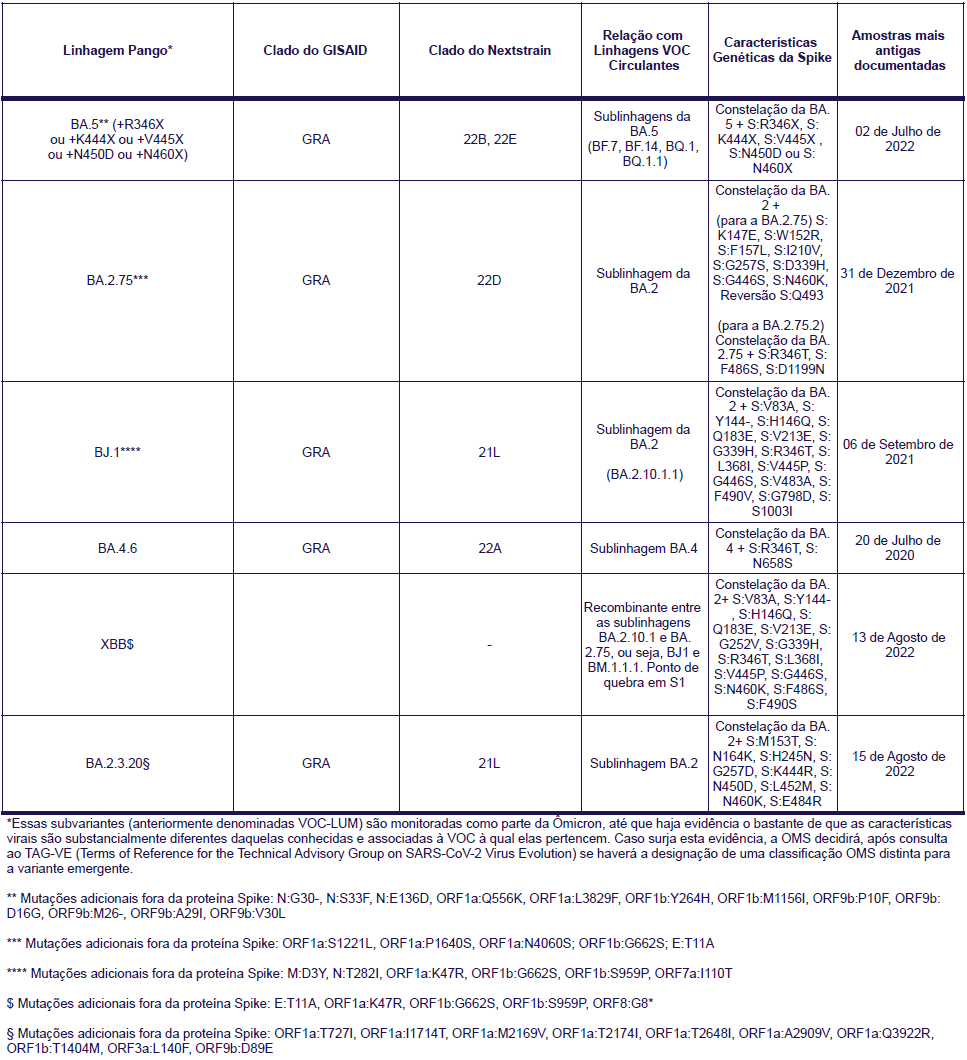
Así, surgen nuevos linajes de diversos organismos a partir de mutaciones, que en su mayoría son perjudiciales para estas entidades. En el caso de los virus, la mayoría de las mutaciones no provocan cambios en la capacidad de dispersión, la infección o la gravedad de la enfermedad. Sin embargo, una minoría de estos cambios puede hacer que el virus se vuelva más transmisible o más mortal.
Los virus como el SARS-CoV-2 cambian más rápidamente que otros microorganismos como las bacterias y los hongos, y se clasifican en linajes distintos por pequeñas diferencias en su material genético, que pueden o no estar asociadas a nuevas características virales. Para entender y estudiar mejor los virus, los científicos han creado un sistema de nomenclatura para los diferentes linajes del SARS-CoV-2, que permite comparar los resultados obtenidos en cualquier región del planeta y detectar qué linajes son más prevalentes y circulan en una zona o en un momento determinado. Hasta ahora se han identificado un conjunto de mutaciones en algunos linajes del coronavirus (SARS-CoV-2) que les permiten ser más transmisibles entre personas, pero no se ha encontrado nada hasta ahora sobre las mutaciones que llevarían a un cuadro más complicado de la enfermedad o incluso a una mayor mortalidad. Dado que los linajes surgen continuamente a medida que el coronavirus infecta a más personas, es evidente la necesidad de controlar la evolución del genoma viral y la prevalencia de los distintos linajes a lo largo del tiempo.
La Organización Mundial de la Salud (OMS) ha definido un sistema de clasificación de estas variantes en función del riesgo que ofrece cada clase para la salud pública, dividiendo en tres categorías algunas de las variantes más extendidas o que contienen mutaciones asociadas a un mayor riesgo de transmisión o de escape del sistema inmunitario (anticuerpos de personas previamente infectadas o vacunadas). Estas categorías son: Variantes preocupantes (VOC), Variantes de interés (VOI) y Variantes bajo vigilancia (VUM).
Según la página de la OMS dedicada a la cuestión de las variantes, los criterios para clasificar las VOC son: cumplir los criterios para la clasificación como VOI, además de estar asociadas de forma demostrable a: un mayor riesgo de relevancia mundial en su transmisibilidad, virulencia y manifestaciones clínicas; o una disminución de la eficacia de las medidas de salud pública.