Thus, new lineages of various organisms arise from mutations, which are mostly detrimental to these entities. In the case of viruses, most mutations do not cause changes in the ability to spread, infect, or the severity of the disease. However, a minority of these changes can lead to the virus becoming more transmissible or more deadly.
Viruses such as SARS-CoV-2 change more rapidly than other microorganisms such as bacteria and fungi, and are classified into distinct lineages by small differences in their genetic material, which may or may not be associated with new viral characteristics. To better understand and study the viruses, the scientists have created a naming system for the different lineages of SARS-CoV-2, which allows them to compare the results obtained in any region of the planet and detect which lineages are more prevalent and circulating in an area or at a given time. So far a set of mutations have been identified in some coronavirus lineages (SARS-CoV-2) that allow them to be more transmissible between people, but nothing has been found so far about mutations that would lead to a more complicated picture of the disease or even higher mortality. Because lineages continuously emerge as the coronavirus infects more people, there is a clear need to monitor the evolution of the viral genome and the prevalence of different lineages over time.
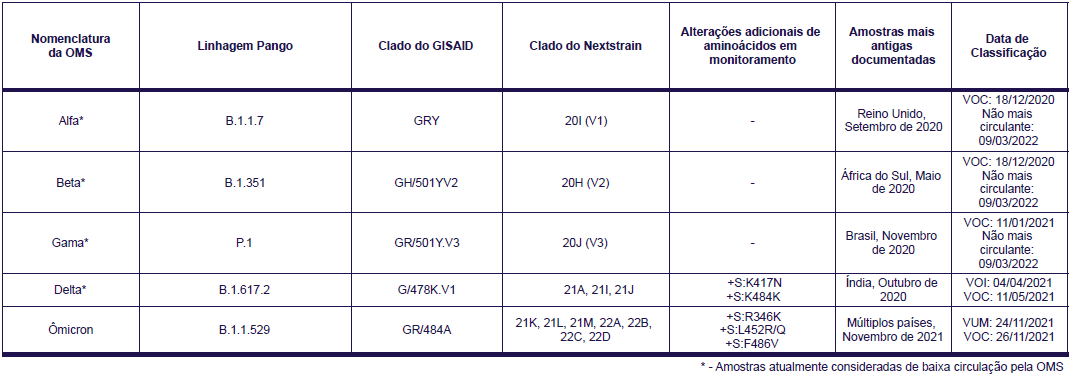
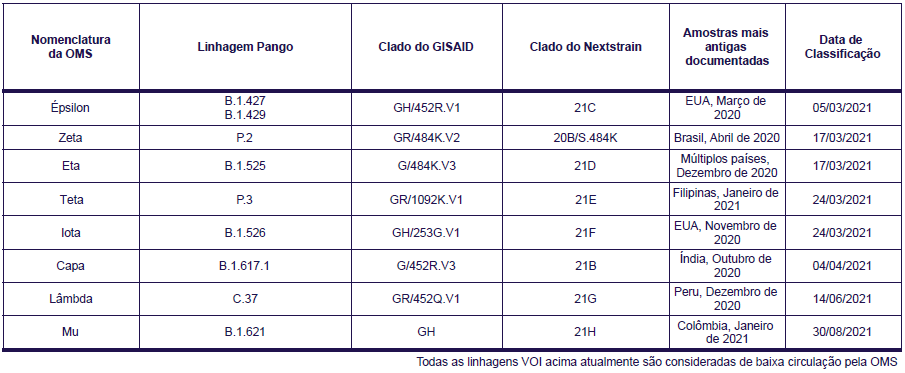
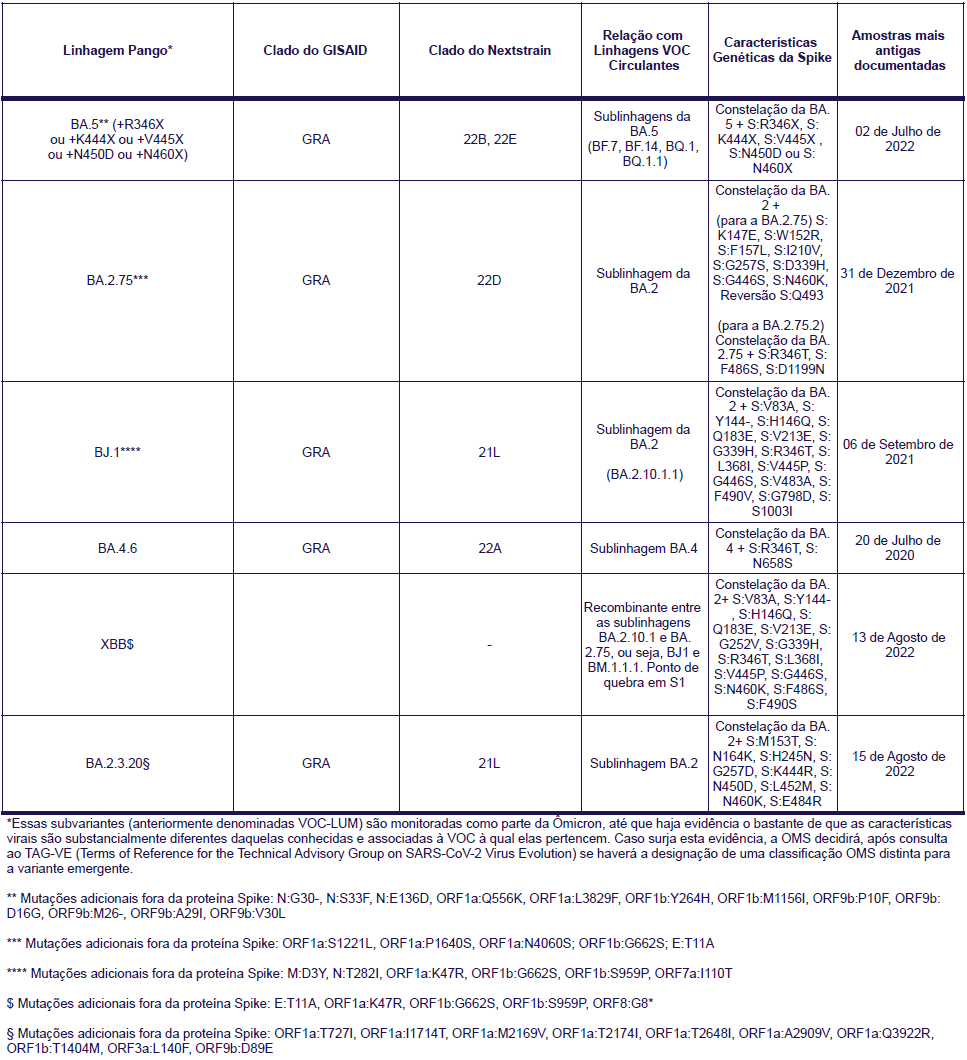
The World Health Organization (WHO) has defined a classification system for these variants according to the risk each class poses to public health, dividing into three categories some of the more widespread or mutated variants associated with a higher risk of transmission or immune system escape (antibodies from previously infected or vaccinated people). These categories are: Variants of Concern (VOC), Variants of Interest (VOI), and Variants Under Monitoring (VUM).
According to the WHO page dedicated to the issue of Variants, the criteria for classification of VOCs are: meeting the criteria for classification as a VOI, in addition to being demonstrably associated with: an increased risk of global relevance in its transmissibility, virulence and clinical manifestations; or a decrease in the effectiveness of public health measures.