Os termos deste Glossário, disponibilizados em ordem alfabética, foram elaborados com base em verbetes encontrados no site e nas publicações da Rede Genômica Fiocruz. O conteúdo desta página está em constante revisão e novos termos serão adicionados ao longo do tempo.
Amplicon
Nome dado a pedaços de DNA amplificados em reações de PCR. Tanto produtos da reação quanto as sequências utilizadas como modelo recebem o nome de “amplicon”.
Análise Bayesiana
A estatística Bayesiana consiste em uma abordagem para o tratamento de dados onde, diferente da estatística clássica (frequentista), o conhecimento anterior a respeito do fenômeno estudado é levado em conta explicitamente nas análises. Esta abordagem pode proporcionar análises mais bem embasadas, por levar em conta modelos construídos com base em dados prévios (por exemplo, de pesquisas realizadas anteriormente) ao invés das distribuições hipotéticas utilizadas em testes estatísticos frequentistas. Do ponto de vista da análise filogenética, a análise Bayesiana envolve a incorporação do conhecimento prévio a respeito de grupos de organismos (ou vírus) para derivar conclusões a respeito do parentesco de novas amostras analisadas — tanto entre si quanto em relação aos organismos já conhecidos previamente.
Ancestral comum
Em biologia evolutiva, um “ancestral comum” é aquele do qual derivam outros organismos. Essa ancestralidade pode se dar em diferentes escalas, de forma a ser possível falar de:
- Grupos inteiros de organismos que se derivam de ancestrais comuns (como no caso das aves, que descendem de dinossauros);
- Espécies de um mesmo grupo que partilham ancestrais comuns. Por exemplo, três das espécies do gênero Panthera, os leões, os leopardos e as onças-pintadas, descendem de um mesmo ancestral, que é diferente da espécie que deu origem aos tigres e aos leopardos-das-neves. Por sua vez, essas duas espécies ancestrais também se derivaram de um mesmo ancestral comum;
- Linhagens dentro de uma mesma espécie, como no caso das linhagens do SARS-CoV-2, causador da COVID-19. A partir de um genoma viral comum, novas linhagens foram derivando pelo acúmulo de mutações genéticas, de forma a ser possível agrupar variantes em grupos de acordo com suas histórias genéticas comuns;
Arbovírus / Arbovirose
As arboviroses são um conjunto de doenças causadas por vírus transmitidos pela picada de artrópodes hematófagos (principalmente insetos de uma gama ampla de gêneros, como os mosquitos Aedes, Culex e Anopheles, mas alguns arbovírus também podem ser transmitidos por espécies de carrapatos e moscas). O grupo dos arbovírus na realidade reúne diversas famílias virais (como Flaviviridae, Orthomyxoviridae e Togaviridae) que, independentemente, adaptaram-se a um ciclo de vida envolvendo vetores artrópodes e hospedeiros vertebrados. A grande maioria dos arbovírus conhecidos têm o genoma tipo RNA, genomas estes que podem ou não ser segmentados em mais de uma molécula, e/ou com fitas simples ou duplas.
Infecções por arbovírus, como a dengue e a febre amarela, têm séculos de registro na história humana. Descrições de casos com sintomas compatíveis com o quadro de dengue, por exemplo, são encontradas em enciclopédias médicas da China, com casos datando de 265 d.C. Com a expansão do meio urbano e avanço de populações humanas sobre áreas naturais, a exposição de pessoas aos ciclos de circulação dos arbovirus foram se tornando mais comuns (pelo maior convívio com os artrópodes que agem como vetores para esses patógenos), com registros mais recentes de surtos e epidemias, além do isolamento e estudo dos vírus em si.
As arboviroses têm se tornado motivo de preocupação maior por parte de órgãos mundiais de saúde como a Organização Mundial de Saúde, devido a um potencial risco aumentado de surtos e epidemias destas doenças associado às mudanças climáticas globais. Períodos de calor mais intenso, e de duração mais prolongada, tendem a aumentar as populações de artrópodes vetores de várias arboviroses, uma vez que a viabilidade de ovos, larvas e adultos é maior no calor. Este fator, somado à ocorrência maior de chuvas e inundações (para o caso de mosquitos) pode levar a uma expansão dos tamanhos populacionais e da área de ocorrência dos vetores de arboviroses, um fator importante para o estabelecimento de ciclos de circulação viral em regiões onde estas viroses não eram comuns.
Bioinformática
Bioinformática é o nome dado às diversas aplicações da computação desenvolvidas para o estudo da biologia. Os usos da bioinformática vão de programas de computador para a análise de sequências genômicas e comparação com sequências parecidas já identificadas até a modelagem tridimensional da estrutura de biomoléculas como proteínas e açúcares. No contexto da Rede Genômica, a bioinformática é utilizada principalmente para analisar e comparar os genomas de diferentes amostras do novo coronavírus SARS-CoV-2, de forma a identificar mutações características das novas variantes e mesmo detectar mutações novas. O uso das ferramentas de modelagem tridimensional de proteínas também está ganhando mais espaço na Rede Genômica, o que permitirá estudar a fundo a proteína Spike do novo coronavírus — que permite sua entrada nas células — e os efeitos de mutações na estrutura e na funcionalidade desta proteína, com possível aumento ou diminuição da infectividade.
Coronavírus
Grupo de vírus (subfamília Orthocoronaviridae) descoberto em 1937, associado principalmente a doenças respiratórias em animais, incluindo a espécie humana. A atual pandemia é causada pelo vírus denominado SARS-CoV-2, devido a sua similaridade com o agente causador da epidemia de SARS (Síndrome Respiratória Aguda Grave; do inglês Severe Acute Respiratory Syndrome) em 2003.
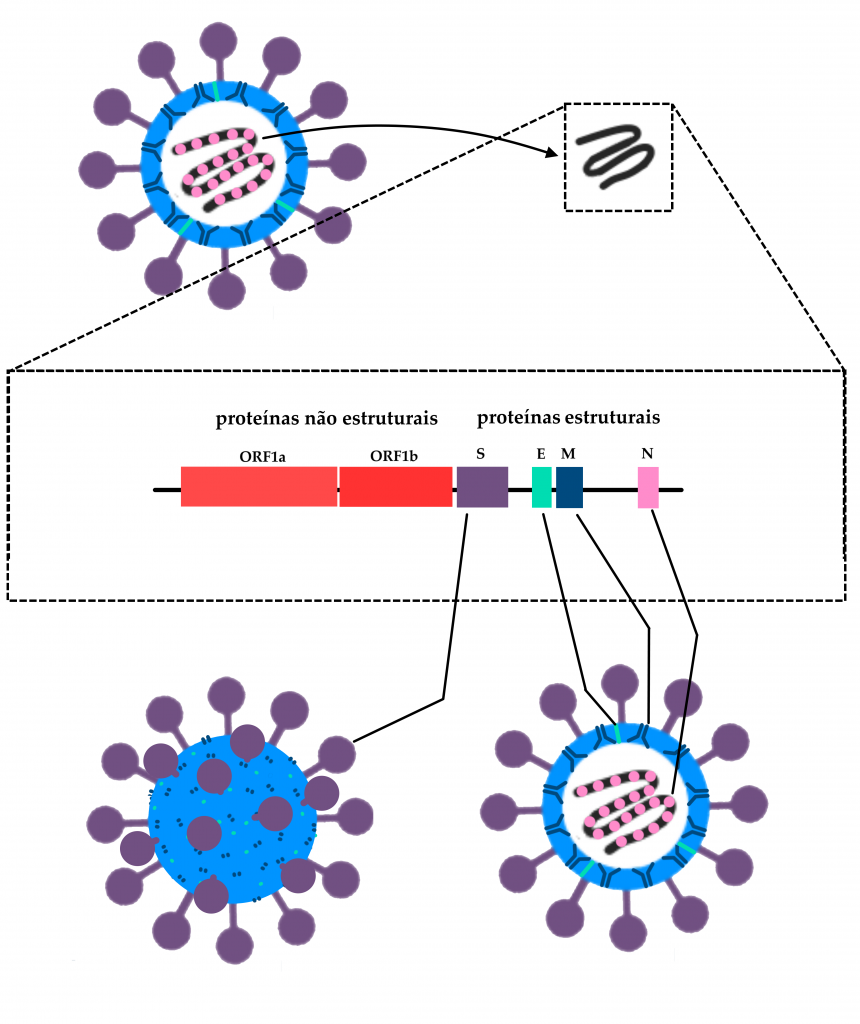
Esquema simplificado da estrutura genômica do SARS-CoV-2, com destaque para os genes que codificam a Glicoproteína Spike (S), a proteína do envelope (E), a proteína da membrana (M) e a proteína que envolve o RNA dentro da partícula viral completa, formando o nucleocapsídeo (N). Também em destaque estão as regiões do genoma denominadas ORF1a e ORF1b, onde estão localizados parte dos genes de proteínas não-estruturais do vírus — ou seja, que não fazem parte da estrutura da partícula viral. Estas proteínas têm funções diversas na infecção, como fazer cópias do RNA viral dentro da célula e redirecionar o metabolismo celular para transformar a célula numa “fábrica de vírus“.
Antes do SARS-CoV-2, foram identificados outros seis coronavírus capazes de causar infecções em seres humanos, sendo quatro destes envolvidos em quadros leves similares a resfriados comuns. Somados a estes quatro coronavírus de perfil mais brando de doença e ao SARS-CoV (ou SARS-CoV-1), causador da epidemia de 2003 caracterizada por um quadro respiratório mais grave, o outro coronavírus causador de doença em humanos é o agente causador da MERS (Síndrome Respiratória do Oriente Médio; do inglês Middle East Respiratory Syndrome), epidemia associada à criação de camelos e a taxas de mortalidade mais altas (37%) do que a SARS e a COVID-19.
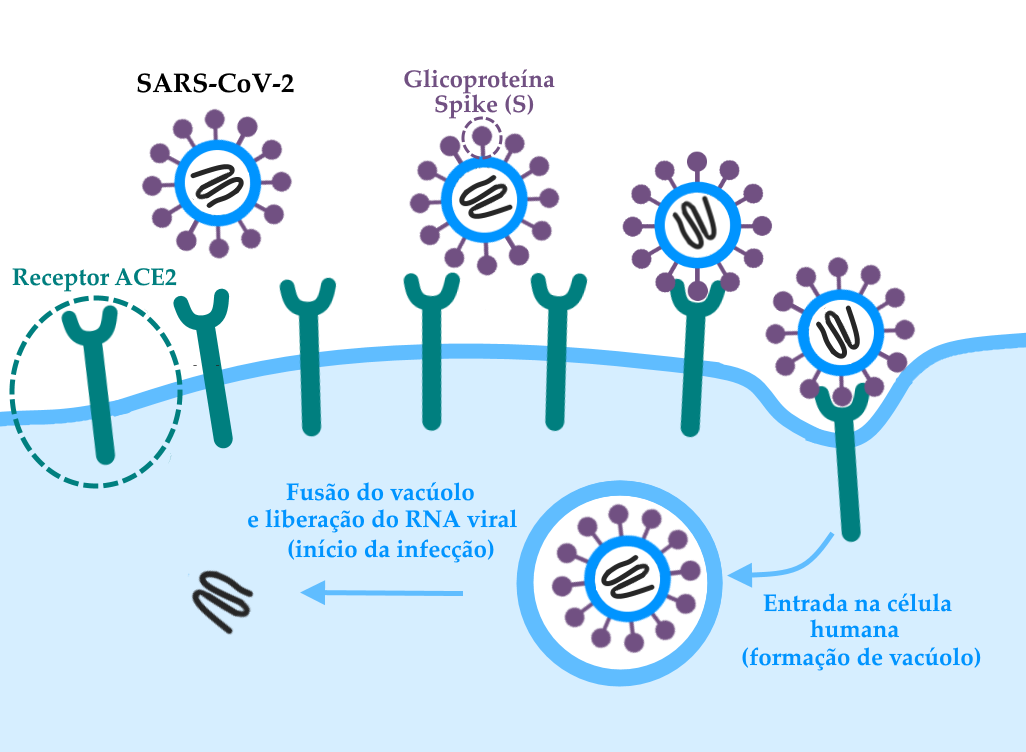
O coronavírus entra nas células a partir da ligação da proteína denominada Spike (ou S), com um domínio que se liga à enzima conversora de angiotensina 2 — proteína que fica na superfície de células humanas e age como “receptor” do vírus — e outro domínio que faz com que a ocorra a fusão entre a membrana da célula e a partícula viral, permitindo a entrada do material genético (RNA) do vírus e o princípio da infecção.
Esquema ilustrando a ligação entre a glicoproteína Spike (proteína S) do SARS-CoV-2 e a Enzima Conversora de Angiotensina 2 (ACE-2) presente em células humanas. Após a ligação, a célula internaliza a partícula viral em um vacúolo, que depois se funde ao envelope viral, liberando o material genético que dará início à infecção.
A hiperinflamação característica da COVID-19 é um dos principais mecanismos patogênicos da doença, resultando em febre e perda de função respiratória, que por sua vez pode levar à necessidade de internação e ventilação mecânica em casos mais graves. O excesso de produção de citocinas — moléculas de comunicação entre células, envolvidas na regulação de mecanismos imunológicos — associadas a quadros inflamatórios está diretamente relacionada à febre e ao comprometimento de tecidos seguido de fibrose, em um fenômeno denominado “tempestade de citocinas”. A tempestade de citocinas está associada a outros quadros virais que têm nos efeitos da hiperinflamação uma parte importante da patogênese, como casos graves de dengue, por exemplo.
Acredita-se que o SARS-CoV-2 tenha se originado em espécies de morcegos e devido a mutações conseguiu infectar e ser transmitido entre outras espécies de mamíferos intermediários, incluindo eventualmente a espécie humana. Análises de amostras circulando entre outras populações animais colocam o pangolim da Malásia como um possível intermediário neste processo de adaptação para outros hospedeiros (também chamado de spillover, do inglês para “transbordar”). A pandemia de COVID-19, com mais de 2 milhões de mortes confirmadas mundialmente ainda em janeiro de 2021, é a maior vivida pela humanidade desde a primeira pandemia de H1N1, de 1917-1918, que ocasionou um total estimado de 50 milhões de mortes globalmente.
COVID-19
Nome dado à doença causada pelo novo coronavírus (denominado SARS-CoV-2). Do inglês, COrona VIrus Disease 19 (por conta do ano de início da pandemia, 2019). A COVID-19 é caracterizada por alta transmissibilidade e, por uma porcentagem de casos assintomáticos ou apresentando sintomas leves. Após um período de incubação de 2 a 14 dias, podem surgir sintomas iniciais pouco específicos (tosse, febre, perda olfativa, dor de garganta, dor de cabeça, fadiga e, eventualmente, diarréia), que podem ser seguidos de desconforto respiratório, hipóxia e inflamação grave do trato respiratório, incluindo os pulmões.
Por ser uma doença ainda muito recente, o conhecimento acerca da COVID-19 ainda precisa de maiores estudos, principalmente em relação aos efeitos de longo prazo, assim como comorbidades decorrentes da infecção pelo SARS-CoV-2, tanto em pessoas sintomáticas como assintomáticas. Em casos mais raros, pode ocorrer encefalite (inflamação cerebral) e o acometimento de outros tecidos, com quadros como epididimite e complicações vasculares aparecendo na literatura científica.
Evolução do genoma viral
Evolução do genoma significa a soma dos processos de: surgimento de diferenças genéticas; acúmulo de mutações; recombinações genéticas que podem ocorrer quando um mesmo paciente é infectado por duas variantes de genéticas diferentes; e, por fim, o processo de seleção natural que atua sobre as mutações genéticas favorecendo aquelas que apresentaram algum tipo de vantagem sobre as outras, por exemplo, uma capacidade infectiva maior.
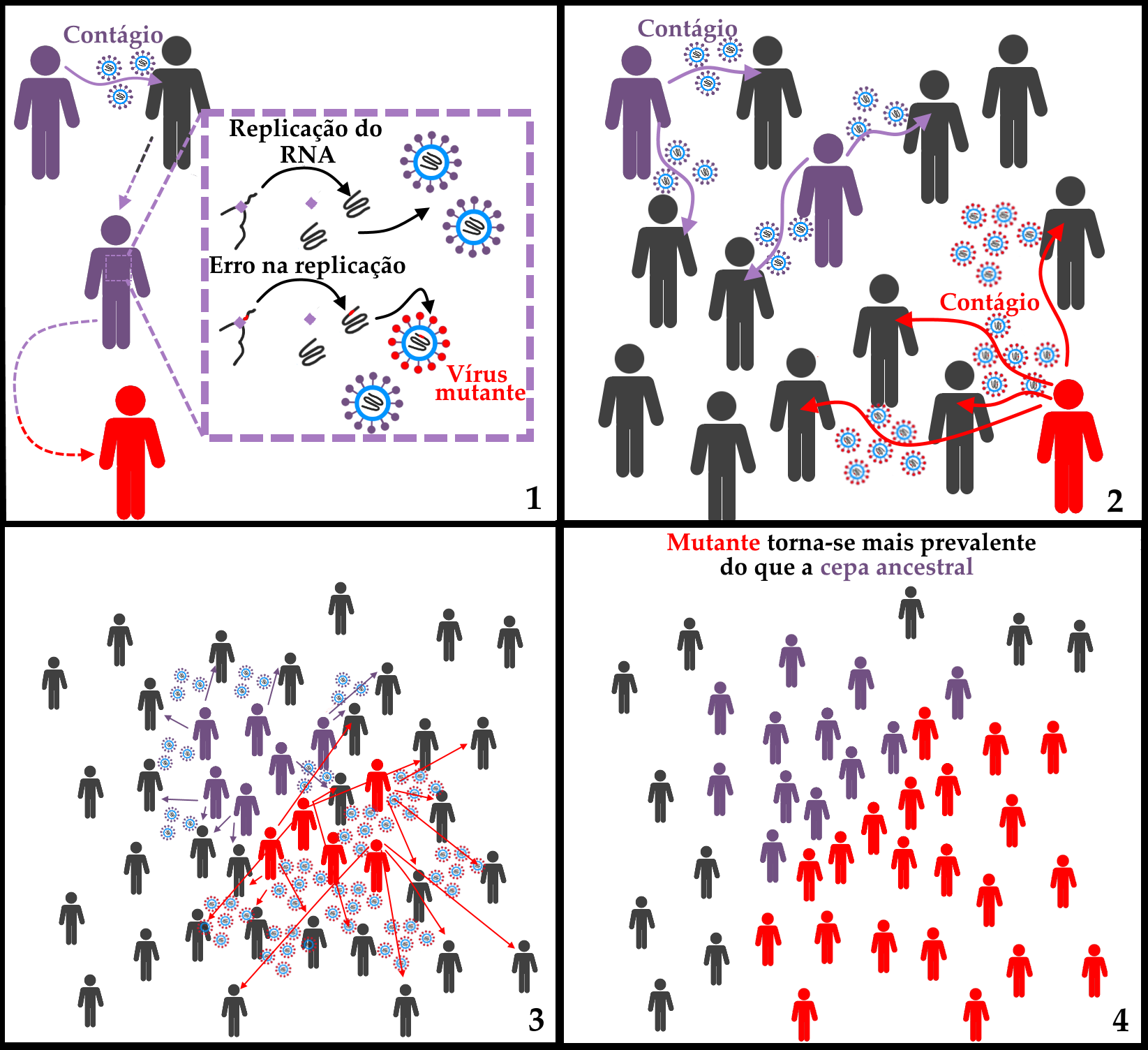
Esquema representando dois processos da evolução viral. No primeiro momento (1), devido a erros aleatórios aos quais o processo de replicação do RNA sempre está sujeito, surge uma amostra mutante. No exemplo hipotético do esquema, a alteração no genoma causa uma mudança na proteína S, que proporciona uma maior infectividade do vírus (2). Aos poucos, a vantagem conferida à maior infectividade (3) faz com que o vírus contendo a mutação torne-se cada vez mais comum. A cada nova geração do vírus, os mecanismos da seleção natural continuam agindo, de forma que a mutante torna-se mais prevalente que sua ancestral (4) devido à vantagem conferida pela mutação.
Acompanhar a evolução de um vírus significa olhar para novas linhagens e como elas diferem entre si, qual a proporção de cada uma na população total da espécie, e como as tendências da epidemia (crescimento, estabilização ou aumento no número de casos) sofrem influência distribuição de diferentes variantes e cepas de um vírus.
A evolução de um vírus não significa necessariamente que ele causa um quadro clínico mais grave, ou que leva a maiores taxas de mortalidade. Na maioria das vezes, as mutações não têm impacto significativo sobre a biologia do vírus.
Febre de Chikungunya
Arbovírus da família Togaviridae identificado pela primeira vez na Tanzânia, transmitido pela picada de mosquitos das espécies Aedes aegypti e Aedes albopictus e causador de um quadro de saúde caracterizado por febre, dor nos músculos e articulações e, em alguns casos, irritação na pele. Os sintomas aparecem tipicamente entre dois e doze dias após o contágio, e duram uma semana, podendo ocorrer um quadro persistente de dor e inflamação nas articulações que dura meses após a eliminação da infecção. O vírus Chikungunya tem genoma baseado em uma fita simples de RNA, e seu ciclo viral se completa após a invasão de células da pele, como fibroblastos e células epiteliais, e macrófagos, que são células do sistema imunológico.
Os primeiros casos de chikungunya registrados no Brasil (e nas Américas como um todo) ocorreram em 2014, e o vírus conseguiu estabelecer uma cadeia de circulação estável no país, com ondas epidêmicas e a manutenção do vírus em mais de uma região do país (como no Rio de Janeiro, Amapá, Amazonas, Roraima e Bahia). Dois diferentes genótipos circulam no território brasileiro: o ECSA (África Leste-Central-Sul) e o Asiático.
Febre de Dengue
Causada por um arbovírus da família Flaviviridae, a dengue foi a segunda doença humana cujo agente etiológico foi identificado como sendo um vírus (após o vírus da febre amarela), em 1907. O vírus da Dengue tem um genoma não-segmentado, baseado em uma fita simples de RNA. A infecção pelo vírus da Dengue se dá após a picada de um mosquito do gênero Aedes infectado. Durante a picada, o mosquito libera partículas virais, que podem infectar células da pele, incluindo as chamadas Células de Langerhans — que são macrófagos do sistema imunológico residentes nos tecidos. Após infectadas, estas células migram para os linfonodos, onde o vírus pode infectar outras células brancas do sangue.
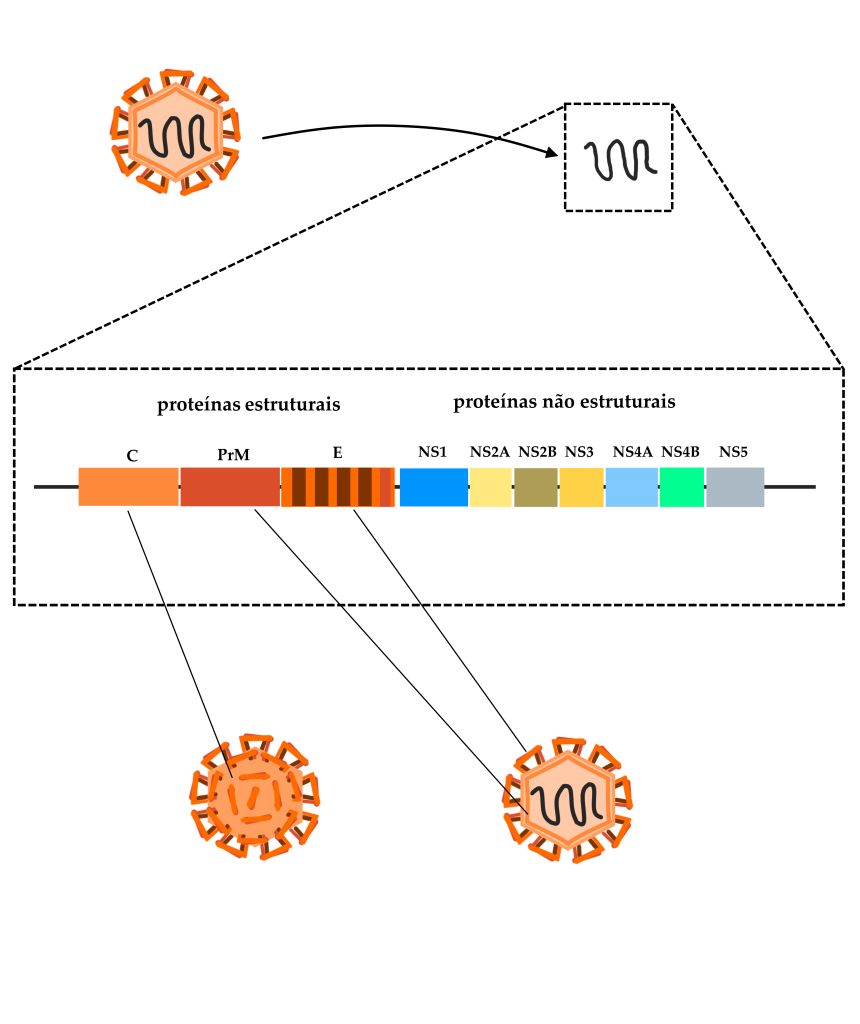 Esquema simplificado da estrutura genômica do vírus da Dengue, com destaque para os genes que codificam a proteína do capsídeo (C), a proteína pré-membrana (PrM), a proteína do envelope (E), além dos genes de proteínas não-estruturais do vírus. As proteínas não-estruturais, apesar de não comporem a estrutura da partícula viral, têm funções diversas na infecção, como fazer cópias do RNA viral dentro da célula e redirecionar o metabolismo celular para a produção de novas partículas.
Esquema simplificado da estrutura genômica do vírus da Dengue, com destaque para os genes que codificam a proteína do capsídeo (C), a proteína pré-membrana (PrM), a proteína do envelope (E), além dos genes de proteínas não-estruturais do vírus. As proteínas não-estruturais, apesar de não comporem a estrutura da partícula viral, têm funções diversas na infecção, como fazer cópias do RNA viral dentro da célula e redirecionar o metabolismo celular para a produção de novas partículas.
Quando sintomática, a dengue tem um quadro clínico caracterizado por febre alta, dor de cabeça, náusea, vômito, dores nas articulações e músculos, podendo os sintomas, em casos graves, serem acompanhados de hemorragia. Os sintomas normalmente aparecem entre 3 e 14 dias após o contágio, desaparecendo entre 2 e 7 dias após seu início. Quadros graves de dengue são caracterizados por uma superativação do sistema imunológico, com um fenômeno chamado de “tempestade de citocinas”, também envolvido na patofisiologia de outras doenças virais graves. Esta superativação imunológica pode levar ainda à síndrome de choque da dengue, que se caracteriza por uma queda drástica na pressão sanguínea e à falência de órgãos. Os quadros hemorrágicos e de síndrome de choque, embora graves, são mais raros do que os casos de febre da dengue.
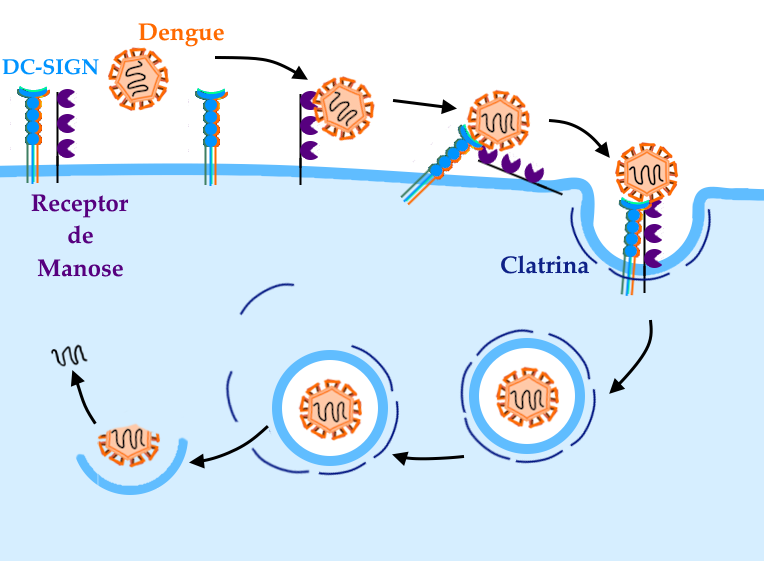 Esquema simplificado das etapas iniciais da infecção pelo vírus da Dengue em um macrófago pela via clássica (existem vias alternativas de invasão celular). O primeiro passo para a infecção de um macrófago é a adesão das proteínas do Envelope e receptores celulares que se ligam a ela. Os mais conhecidos destes receptores são o DC-SIGN e o Receptor de Manose, que se ligam ao vírus e sinalizam para a célula que existe algo a ser fagocitado. A fagocitose se dá pelo envolvimento do vírus em uma invaginação de membrana chamada lisossomo, que é envolvido por proteínas chamadas clatrina, que o estabilizam. Após a acidificação do lisossomo, que é um mecanismo da própria fagocitose para a quebra de patógenos ou moléculas estranhas, o capsídeo viral se funde à célula e libera o RNA viral no citoplasma, iniciando a infecção propriamente dita.
Esquema simplificado das etapas iniciais da infecção pelo vírus da Dengue em um macrófago pela via clássica (existem vias alternativas de invasão celular). O primeiro passo para a infecção de um macrófago é a adesão das proteínas do Envelope e receptores celulares que se ligam a ela. Os mais conhecidos destes receptores são o DC-SIGN e o Receptor de Manose, que se ligam ao vírus e sinalizam para a célula que existe algo a ser fagocitado. A fagocitose se dá pelo envolvimento do vírus em uma invaginação de membrana chamada lisossomo, que é envolvido por proteínas chamadas clatrina, que o estabilizam. Após a acidificação do lisossomo, que é um mecanismo da própria fagocitose para a quebra de patógenos ou moléculas estranhas, o capsídeo viral se funde à célula e libera o RNA viral no citoplasma, iniciando a infecção propriamente dita.
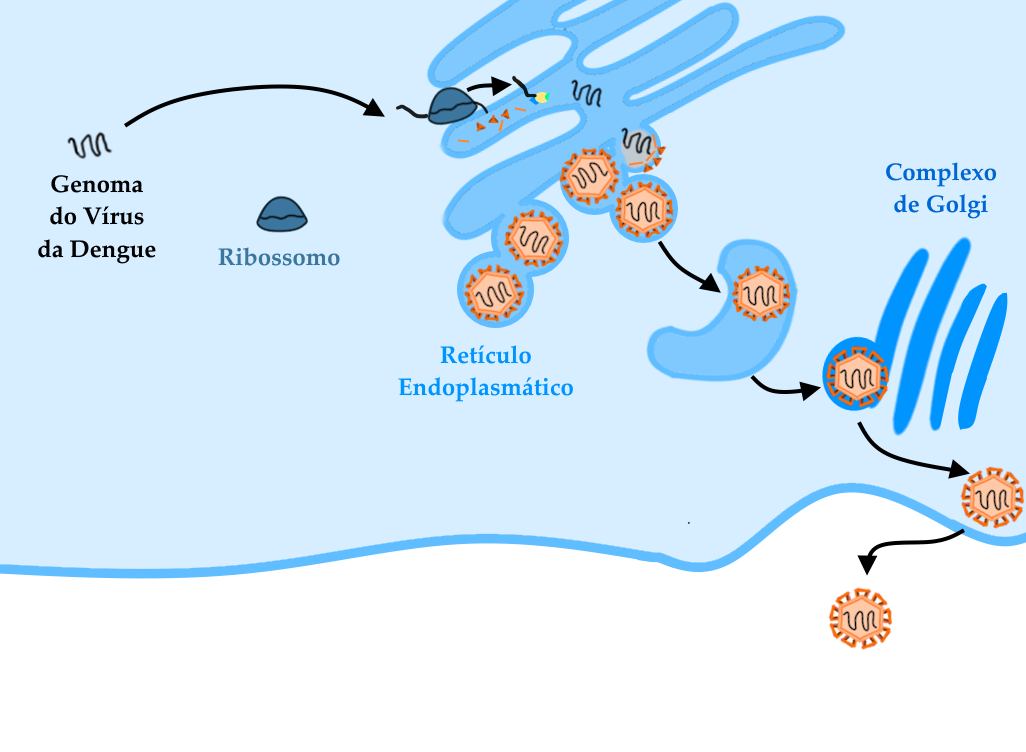 Esquema simplificado das etapas da infecção pelo vírus da Dengue após a liberação do genoma viral no citoplasma da célula. O RNA viral é traduzido pelos ribossomos, muitos dos quais já estão próximos ao retículo endoplasmático. Uma vez liberadas no retículo endoplasmático, as proteínas virais serão modificadas por enzimas do retículo, tornando-se mais próximas de sua forma final e ativa. Também no retículo, as proteínas não estruturais irão agir, tanto alterando o metabolismo da célula quanto promovendo a replicação do genoma do vírus. Após a montagem de capsídeos virais (ainda imaturos) no retículo endoplasmático, as partículas virais passarão pelo complexo de Golgi, onde haverá novas alterações de suas proteínas e a liberação de vírus maduros e funcionais. Estes vírus serão posteriormente eliminados da célula hospedeira onde, uma vez na corrente sanguínea, poderão infectar outras células ou mesmo serem ingeridos por mosquitos.
Esquema simplificado das etapas da infecção pelo vírus da Dengue após a liberação do genoma viral no citoplasma da célula. O RNA viral é traduzido pelos ribossomos, muitos dos quais já estão próximos ao retículo endoplasmático. Uma vez liberadas no retículo endoplasmático, as proteínas virais serão modificadas por enzimas do retículo, tornando-se mais próximas de sua forma final e ativa. Também no retículo, as proteínas não estruturais irão agir, tanto alterando o metabolismo da célula quanto promovendo a replicação do genoma do vírus. Após a montagem de capsídeos virais (ainda imaturos) no retículo endoplasmático, as partículas virais passarão pelo complexo de Golgi, onde haverá novas alterações de suas proteínas e a liberação de vírus maduros e funcionais. Estes vírus serão posteriormente eliminados da célula hospedeira onde, uma vez na corrente sanguínea, poderão infectar outras células ou mesmo serem ingeridos por mosquitos.
Registros em enciclopédias médicas da Dinastia Jin descrevem casos clínicos com sintomas compatíveis com a dengue na China antiga (226 a 420 d.C). Os primeiros surtos modernos da doença de que se tem registro ocorreram em 1779. A doença é endêmica em mais de 100 países — em geral, em territórios na zona tropical) e sua prevalência global dá sinais de estar aumentando, em decorrência das mudanças climáticas.
O vírus da Dengue pode ser classificado em quatro sorotipos (DENV-1, DENV-2, DENV-3 e DENV-4), com os quatro circulando em alguma medida no território brasileiro. Estes sorotipos podem ainda ser subdivididos em diferentes genótipos, o que também tem consequências epidemiológicas. A preocupação atual entre os anos de 2023 e 2024 é a detecção de amostras pertencentes a um genótipo do DENV-3 recém-reintroduzido no país que aparentemente conseguiu se estabelecer no cenário epidemiológico, o que pode levar a surtos e epidemias de maior impacto devido ao fato de que, nas últimas décadas, nenhuma linhagem do DENV-3 teve circulação expressiva no país. A falta de exposição recente ao sorotipo 3 do vírus da Dengue pode significar uma baixa memória imunológica na população, o que leva a uma maior vulnerabilidade do povo brasileiro a este genótipo, denominado DENV-3-GIII-American-II.
Filogenética
O estudo das relações de parentesco evolutivo entre organismos — incluindo linhagens virais — a partir da análise de seus genomas tem o nome de “filogenética”. A filogenética é uma forma de organização de formas de vida e outras entidades biológicas de acordo com características e, anteriormente à descoberta do material genético e do desenvolvimento de ferramentas para seu estudo, era realizada pela comparação de características dos organismos, como estruturas de mesma origem — por exemplo, os braços humanos e as nadadeiras laterais de golfinhos, que surgem a partir das mesmas estruturas durante o desenvolvimento embrionário. Mais recentemente, as características comparadas são, em geral, genéticas, ou seja, a estrutura e funcionalidade de genes em comum entre diferentes entidades biológicas.
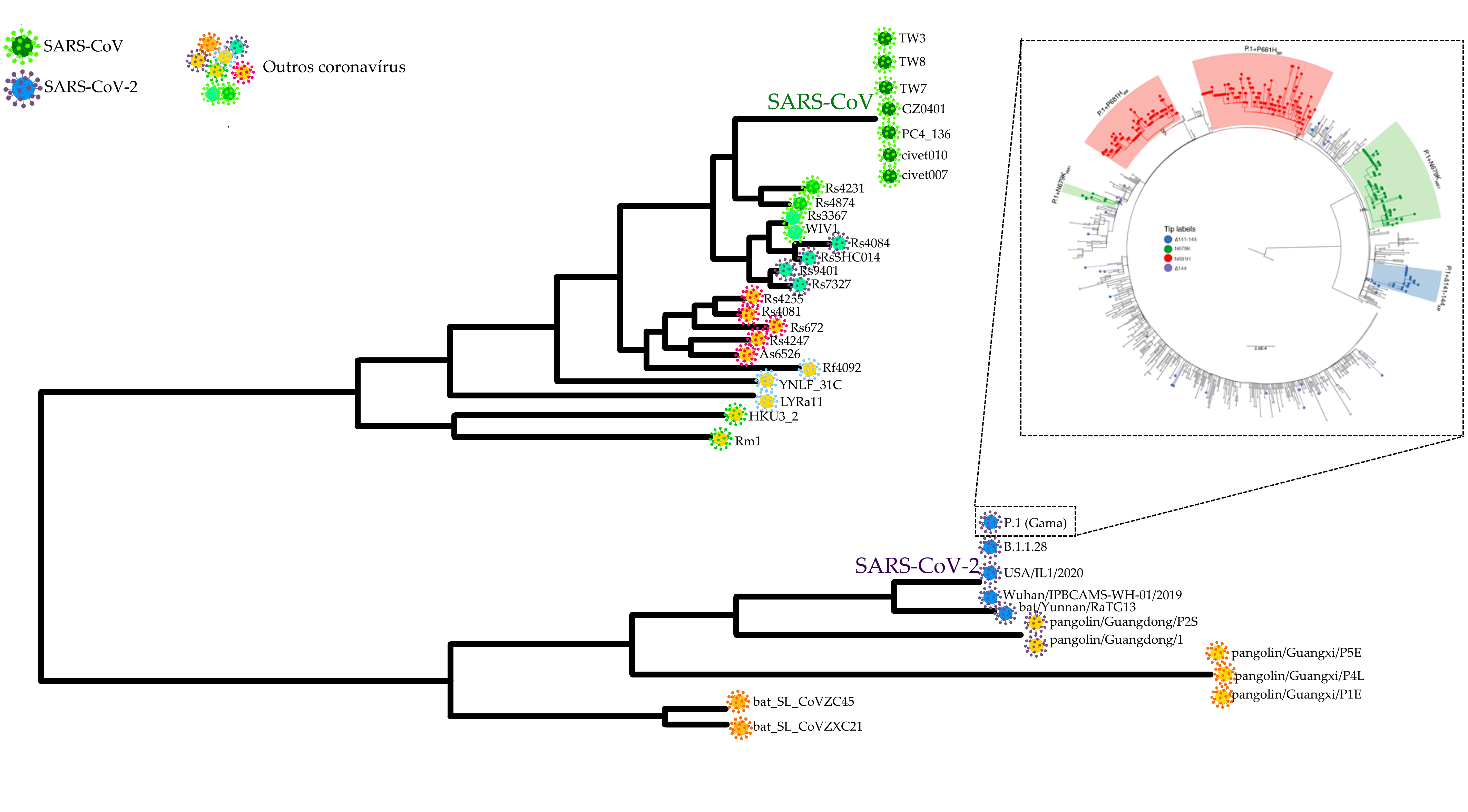
Esquema representando as relações de parentesco entre diferentes amostras de coronavírus, incluindo os causadores da epidemia de SARS na Ásia entre 2002 e 2004 e da pandemia de COVID-19. O esquema maior foi adaptado de Lauxmann et al. (2020), enquanto o esquema detalhando a evolução de descendentes da Variante Gama (P.1) é uma reprodução do esquema elaborado por Naveca et al. (2021) para a postagem no fórum de especialistas Virological.Org.
Gene
Genes são trechos do código genético que, quando lidos e ativados (ou “expressos”), geram algum efeito na célula. O efeito mais clássico de um gene é a fabricação de uma proteína por parte de um intrincado maquinário celular, e a forma e a função desta proteína serão altamente dependentes da estrutura do gene que a originou. Genes podem ainda promover ou silenciar a expressão de outros genes, assim como, no caso de genomas baseados em DNA, dar origem a moléculas de RNA que terão funções efetivas dentro da célula. Quando, durante o processo de duplicação do genoma, ocorre uma alteração na estrutura de um gene, este evento é chamado de “mutação”.
Genética (campo de estudo)
A genética é o estudo da hereditariedade. De forma mais direta, a genética estuda as estruturas do material genético de seres vivos e entidades biológicas como vírus, na intenção de entender como as sequências dão origem às características dos mesmos. O estudo da genética pode ser focado em escalas e objetivos diversos, desde estudar um único gene e a relevância de seu produto (o efeito quando esse gene é expresso, ou “ativado”) até o estudo de como combinações de genes de uma geração podem influenciar as características de seus descendentes.
Genoma
Em contraste à genética, o objetivo da genômica é o estudo dos genes interagindo entre si para compor uma rede integrada de sequências que tendem a atuar de forma coordenada. Em sua totalidade, o genoma de um organismo ou de um vírus possui múltiplos genes. Em organismos de genética mais complexa, genes podem se organizar em grupos que são expressos ou silenciados em conjunto, além de sequências que regulam a intensidade da expressão de outros genes. Além de estudar as sequências de genes individuais, o estudo do genoma envolve a compreensão de todas essas interações entre eles, e de como alterações neste balanço, devido a mudanças no ambiente ou a mutações nas sequências em si, podem impactar positiva ou negativamente os organismos.
Genoma de Alta Qualidade (<1% de N)
Sequência genômica resultante de sequenciamento preciso, na qual há pouca incerteza a respeito dos nucleotídeos em cada posição do genoma. Quando há incertezas no sequenciamento, o software associado ao sequenciador registra “N” na posição da leitura ambígua de nucleotídeos. Uma porcentagem de N inferior a 1% significa que mais de 99% da sequência genômica é conhecida e fruto de um sequenciamento confiável.
GISAID
O GISAID (do inglês para “Iniciativa Global para o Compartilhamento de Dados sobre a Influenza Aviária”) é uma iniciativa de cooperação científica internacional criada em 2008 para tornar mais colaborativos e globais os esforços de monitoramento e desenvolvimento de estratégias de controle das linhagens em circulação dos vírus Influenza (gripe).
Com uma estrutura de bancos de dados e contato entre instituições de pesquisa envolvidas no estudo de genômica viral, o GISAID expandiu o escopo de suas atividades para incluir o novo coronavírus (SARS-CoV-2) e suas linhagens nas atividades de monitoramento e cooperação. O intuito desta expansão é o de permitir um combate coordenado e mais eficiente do espalhamento da doença através da compreensão de como a epidemia/pandemia se desenvolve, assim como o monitoramento quase em tempo real das linhagens do vírus. A Rede Genômica Fiocruz está inserida na cooperação internacional do GISAID, e está gerando e compartilhando dados sobre a COVID-19 no Brasil.
Imunocomprometida
Pessoa que, por qualquer motivo, tenha funcionamento reduzido do sistema imunológico. Dentre os principais motivos para comprometimento do funcionamento da imunidade estão a desnutrição, tratamentos médicos como o uso de corticosteróides ou a quimio e a radioterapia, doenças como HIV/AIDS, leucemias e aplasias medulares, além de situações passageiras, como efeitos do estresse prolongado e a supressão da imunidade naturalmente associada à gravidez.
Infecção por transbordamento
Considerado pelos autores de um artigo de revisão de 2017 como a característica definitiva de patógenos que causam infecção em seres humanos a partir de outras espécies de animais vertebrados (infecções estas conhecidas como “zoonoses”), o transbordamento é o processo complexo no qual um agente causador de doenças em uma espécie ou grupo biológico (como aves, mamíferos ruminantes, etc) consegue se adaptar a outro grupo biológico de hospedeiros. O processo de transbordamento para outros hospedeiros permite ao patógeno causar infecções / epidemias nesta nova espécie ou grupo de espécies, e mesmo tornar-se endêmico em suas populações. O mesmo artigo argumenta que, para haver o transbordamento, é necessário o alinhamento de diversos fatores, como a dinâmica do patógeno na população do hospedeiro original, a dinâmica de contato entre seres humanos e o hospedeiro original (ou vetores da doença), características do patógeno no que diz respeito a sua viabilidade fora do hospedeiro, e características individuais da interação do patógeno com o novo hospedeiro (como a imunidade e a compatibilidade molecular).
Influenza / Influenzavírus
Influenza é o nome dado à doença causada pelos Influenzavírus, caracterizada por sintomas respiratórios como tosse e coriza, febre, dores no corpo e eventualmente sintomas gastrointestinais. Os Influenzavírus são um grupo de vírus respiratórios que atualmente compreende quatro espécies de vírus, Influenza A, Influenza B, Influenza C e Influenza D, cada uma das espécies sendo a única representante de seu respectivo gênero. Todos os vírus Influenza pertencem à família Orthomyxoviridae.
Em contraste ao coronavírus pandêmico SARS-CoV-2, que é o primeiro da família dos coronavírus a causar uma pandemia na espécie humana de que se tem registro na história, a relevância da Influenza para a humanidade remonta à era antiga, como evidenciado pela descrição da doença por Hipócrates no Livro das Epidemias, em 412 a.C., denominada “febre de Perinto”.Já em tempos modernos, alguns eventos epidêmicos e pandêmicos foram bem registrados, sendo provavelmente o mais icônico a pandemia de Influenza A H1N1 de 1918, que causou um número estimado de 50 milhões de mortes em um intervalo de aproximadamente dois anos.
Os sintomas da Influenza sazonal, conforme descrito no site da Organização Mundial de Saúde, são caracterizados por um desenvolvimento súbito da chamada “síndrome gripal”, com febre, tosse (normalmente seca), dor de cabeça, dor muscular e nas articulações, mal estar intenso, dor de garganta e coriza. A tosse pode ser grave e durar duas ou mais semanas. A maior parte das pessoas se recupera em aproximadamente uma semana, sem necessidade de intervenção médica. Mas pessoas em alto risco podem desenvolver quadros graves ou mesmo ir a óbito.
O quadro grave comumente associado à Influenza (mas que pode também ser causado por outros vírus, como o próprio SARS-CoV-2) é a chamada Infecção Respiratória Aguda Grave (IRAG ou SARI), caracterizada por uma inflamação exacerbada no trato respiratório, incluindo dano ao epitélio pulmonar e febre persistente, que demandam hospitalização para monitoramento do estado de saúde do paciente, manejo dos sintomas (com possível necessidade de oxigenação suplementar) e monitoramento para prevenção e combate a infecções bacterianas secundárias (também chamadas de infecções oportunistas, que podem se estabelecer no trato respiratório danificado pelo processo inflamatório). Estima-se que anualmente 400 mil pessoas morram em decorrência da Influenza ao redor do mundo, em situações normais (ou seja, sem eventos pandêmicos ou epidemias de larga escala).
Dos quatro vírus Influenza, apenas os Influenza A, B e C causam doenças em seres humanos. A Influenza A é primariamente um vírus com circulação em aves, mas algumas linhagens deste vírus são capazes de circular entre hospedeiros mamíferos, incluindo a espécie humana. A Influenza B é um vírus primariamente humano, mas há evidências da circulação eventual entre porcos e focas. Já a Influenza C também tem como hospedeiro e reservatório principal a espécie humana, mas também já foi isolado de cães e porcos. Por fim, a Influenza D é um vírus que até o momento só teve circulação evidenciada entre porcos e ruminantes como o gado bovino e caprino.
O genoma dos Influenza A é dividido em oito segmentos de RNA chamados de “cadeia negativa”, ou seja, precisam ser replicados para geração de fitas complementares (cadeia positiva) para que os genes sejam traduzidos. Desta maneira, o RNA do influenza em si possui as sequências complementares às dos genes funcionais.
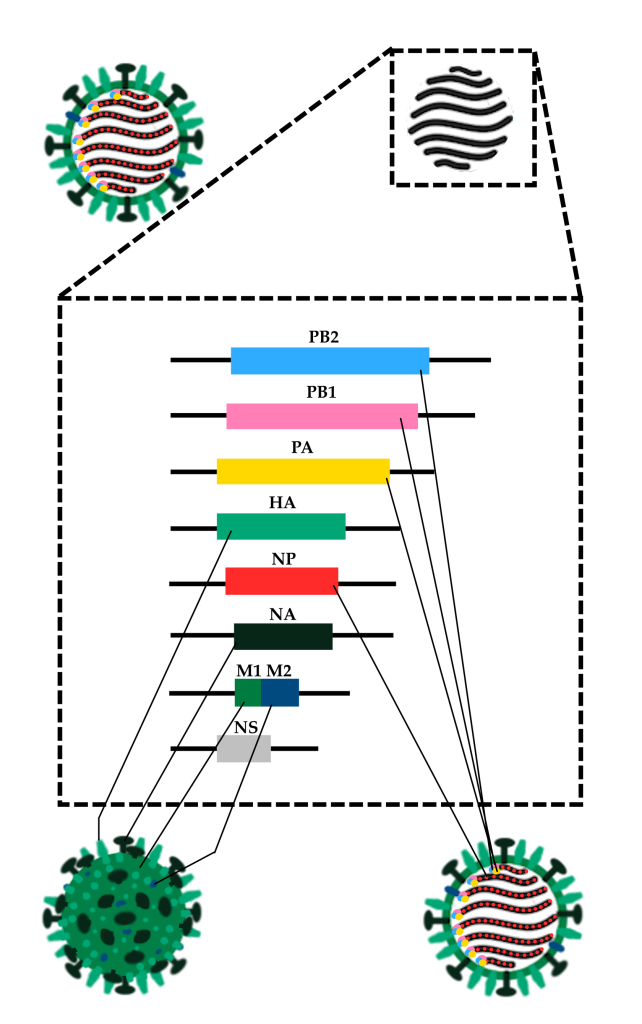
Esquema ilustrando a estrutura do vírus Influenza A, com destaque para as principais proteínas das partículas virais e os respectivos genes que as codificam. PB1, PB2 e PA: Codificam para três subunidades da RNA-polimerase viral, responsável por fazer cópias complementares do RNA viral (que serão traduzidas para outras proteínas ou novamente copiadas para gerar genomas virais completos); HA: Hemaglutinina, proteína responsável pela adesão do vírus à célula hospedeira (através de ligação a receptores ricos no aminoácido “ácido siálico”), e pela fusão da membrana celular ao envelope viral, importante para a liberação do material genético e das proteínas virais na célula hospedeira; NP: Nucleoproteína, que se liga aos nucleotídeos do genoma do Influenza, encapsulando o RNA de fita negativa e sendo um fator importante para o transporte do material genético para o núcleo; NA: Neuraminidase, uma importante enzima para a liberação de partículas virais recém-produzidas em células-hospedeiras, uma vez que a neuraminidase quebra proteínas ricas em ácido siálico, liberando as partículas para infectar novas células; M1 e M2: Proteínas de matriz 1 e 2. A proteína M1 forma uma matriz localizada sob o envelope viral, enquanto a proteína M2 é um importante canal de íons, importante para alterar o pH dentro das partículas virais durante o processo de invasão das células, etapa esta que precede a fusão de membranas; NS: proteína não-estrutural;
A nomenclatura HxNx (por exemplo Influenza A H1N1 ou H3N2) se baseia em duas importantes proteínas de superfície do vírus, a Hemaglutinina e a Neuraminidase, sendo o número correspondente às variações conhecidas das respectivas proteínas. Estas variações são fruto de alterações genéticas, que resultam em diferentes perfis imunogênicos — ou seja, anticorpos que se ligam e neutralizam a Hemaglutinina tipo 1 terão um potencial de neutralização reduzido em relação à Hemaglutinina tipo 3.
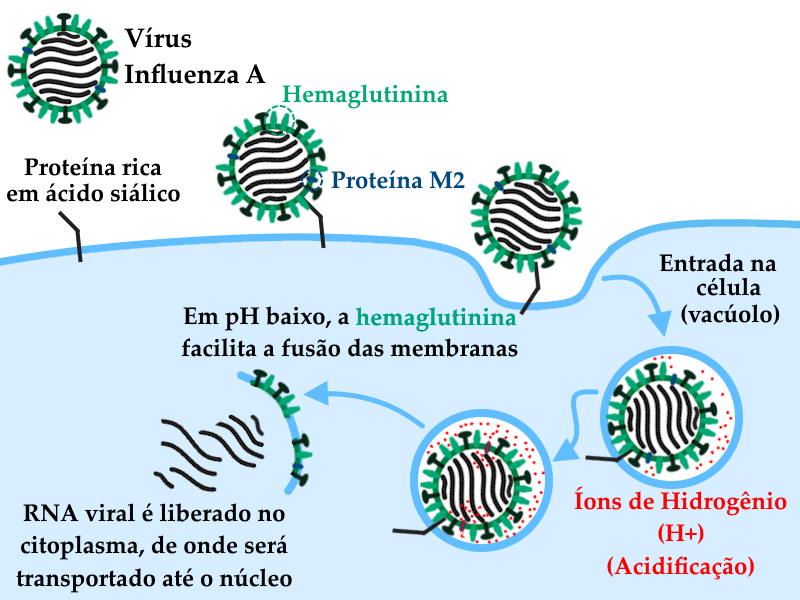
A hemaglutinina tem um papel importante na invasão de células hospedeiras pelo vírus Influenza, pois é a principal molécula responsável pela adesão do vírus à membrana plasmática. Os receptores que se ligam à hemaglutinina são proteínas ricas em ácido siálico, um aminoácido. A ligação do vírus influenza a estes receptores resulta na internalização do vírus em vacúolos de membrana, conforme ilustrado no esquema abaixo.
Esquema ilustrando a adesão e invasão do vírus Influenza A em células hospedeiras. A primeira etapa da invasão é a ligação entre a glicoproteína viral hemaglutinina e proteínas de superfície da célula hospedeira ricas no aminoácido ácido siálico. Após a ligação do vírus a estas moléculas que agem como receptores, mecanismos da própria célula levam à internalização da partícula viral. Uma célula não tem como saber que o que está internalizando em um vacúolo é um vírus. Como ocorre em muitos processos celulares, o vacúolo é acidificado pela célula, com a liberação de íons de hidrogênio, que vão tornando o interior do vacúolo mais ácido do que o citoplasma da célula. A proteína M2 bombeia parte dos íons hidrogênio para dentro da partícula viral, o que é importante para que haja uma acidificação das estruturas do vírus. Esta acidificação (também chamada de “diminuição do pH”) leva a uma mudança na hemaglutinina que, por sua vez, leva a uma facilitação da fusão entre a membrana do vacúolo celular e a membrana do envelope viral. Com a fusão das membranas, o genoma viral é liberado na célula, de onde ele será transportado até o núcleo, que é a região da célula onde começa a replicação viral.
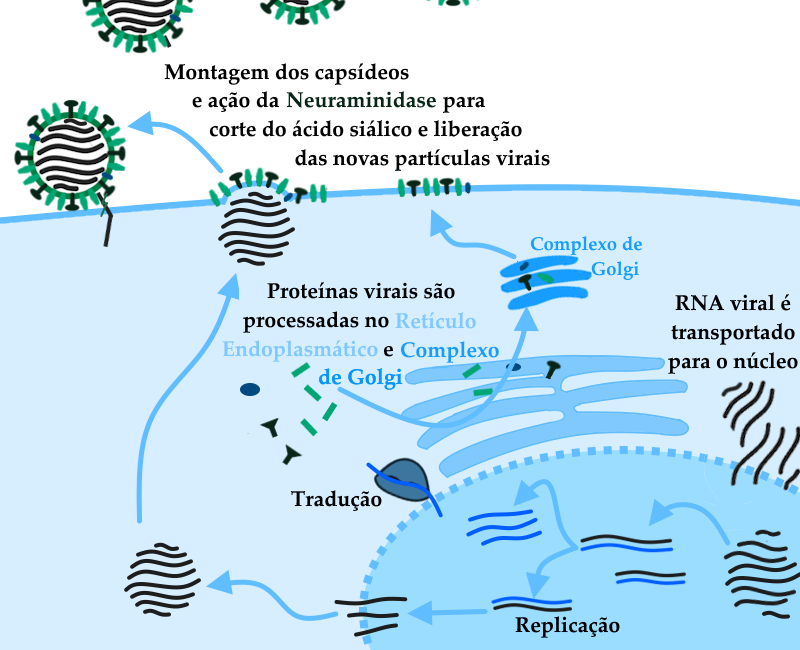
A outra proteína importante para a classificação de linhagens dos vírus Influenza é a Neuraminidase, importante no processo de liberação de novas partículas virais produzidas por uma célula infectada. A Neuraminidase é uma enzima que “corta” trechos de proteínas que possuem ácido siálico, de forma a liberar as partículas virais para seguir com o processo de infecção em outras células do organismo, ou serem transmitidas para outros potenciais hospedeiros.
Esquema representando as etapas do ciclo de replicação viral. Após serem liberados no citoplasma da célula hospedeira, os oito fragmentos de RNA do Influenza são transportados para o núcleo, onde a RNA polimerase do vírus irá agir para geração dos RNAs mensageiros virais — ou seja, as fitas complementares que serão lidas pelos ribossomos da célula para a síntese das proteínas de novas partículas virais. Estas proteínas serão processadas no Retículo Endoplasmático e Complexo de Golgi, processamento este importante para que tenham sua forma final, capaz de realizar suas respectivas atividades biológicas. A partir dos RNAs mensageiros também serão copiadas novas moléculas de RNA viral no núcleo da célula, que também farão parte das novas partículas virais. Após a montagem das partículas novas, estas são liberadas pela ação da Neuraminidase viral, que corta os receptores ricos em ácido siálico liberando o vírus para infectar novas células e hospedeiros.
Por serem peças-chave no processo de infecção viral, a Hemaglutinina e a Neuraminidase são importantes alvos para o desenvolvimento de medicamentos que bloqueiem sua ação. Portanto, são proteínas que têm destaque tanto do ponto de vista de classificação dos vírus em termos de sorotipo quanto em termos de pesquisa e desenvolvimento farmacológico.
Atualmente, temos quatro linhagens principais de influenza circulando sazonalmente. São elas as Influenza A: H3N2 e H1N1 pandêmica; e as linhagens de Influenza B: Victoria e Yamagata. A origem da Influenza A H3N2 remonta ao ano de 1968, quando emergiu em Hong Kong uma linhagem que carregava algumas das mutações anteriormente detectadas na Influenza Aviária de Baixa Patogenicidade e a H2N2 circulante entre seres humanos desde a década anterior. Algumas linhagens de Influenza A H1N1 já emergiram na história humana recente, sendo uma delas a responsável pela pandemia de 1918, outra linhagem envolvida em um surto em 1977 na Rússia e, mais recentemente, a linhagem responsável pela pandemia de 2009, o primeiro evento pandêmico do Século XXI. Já os vírus sazonais de Influenza B , Victoria e Yamagata, são linhagens que divergiram entre si na década de 1970, diferenciando-se em “Yamagata-like” e “Victoria-like” com base nas diferenças nos genes da hemaglutinina e neuraminidase.
Como os vírus Influenza têm o genoma segmentado, é comum em casos de coinfecção um “embaralhamento” dos segmentos do genoma, gerando linhagens que combinem trechos do genoma de duas ou mais linhagens parentais. Este tipo de evento está envolvido no surgimento de linhagens que combinam as características já selecionadas em outras linhagens, e têm relevância pelo fato de estarem eventualmente por trás do surgimento de variantes de maior potencial pandêmico, maior gravidade de sintomas ou maior resistência a antivirais.
Letalidade
Número de mortes na porção afetada de uma população em um determinado intervalo de tempo. Este indicador, diferente da Mortalidade, leva em conta apenas a parcela da população acometida pela doença (ou fator de risco), e não a população integral. Desta forma, a Letalidade é uma medida do risco de uma causa para a totalidade de pessoas afetadas — no caso da COVID-19, por exemplo, o indicador representa a chance de uma pessoa infectada ir a óbito, enquanto a Mortalidade coloca estes óbitos em perspectiva em relação a toda a população.
Linhagens (vírus)
Conjunto de vírus geneticamente relacionados que descendem de um ancestral comum. Uma linhagem deve possuir mutações que a diferenciem de outras variantes do vírus. Estas mutações não precisam modificar nenhuma característica biológica do vírus (ex.: transmissibilidade ou potencial de causar doenças). Para ser denominada como linhagem é necessário que a mesma possua uma relevância epidemiológica, ou seja, esteja circulando em uma grande população.
Material genético
Na biologia, a hereditariedade — ou seja, a transmissão de características de uma geração para a outra — se dá a partir do material genético. É costume popular falarmos que algo está no DNA, mas alguns vírus, como os coronavírus e o HIV, por exemplo, têm seu material genético baseado em RNA. Este tipo de molécula de ácido nucleico também está presente em nossas células, mas está envolvida com processos do metabolismo celular e fluxo da informação genética, atuando, por exemplo, no processo de produção de proteínas.
O material genético viral, ao ser inserido na célula hospedeira, logo é lido pela própria célula e dá origem a proteínas virais que começam o processo de redirecionar o metabolismo celular para a produção de novos vírus. As novas cópias do material genético viral também são feitas com base no material que entra nas células, e é por conta deste processo de fazer cópias com base no “molde” genético dos vírus que eventuais mutações podem ser passadas adiante.
Morbidade
Em epidemiologia, o conceito de morbidade difere daquele utilizado normalmente na clínica — onde o termo significa a gravidade dos sintomas de uma doença, ou o quanto ela debilita um paciente. No contexto epidemiológico, “morbidade” denota a proporção de pessoas afetadas ou portadoras de uma condição, em relação à população geral estudada, em um determinado local e momento. Por exemplo, se em uma vila de 1000 habitantes, 100 pessoas são afetadas por uma doença, a morbidade desta doença no local é de 0,1 ou 10%.
Mortalidade
Número de mortes em uma população em um determinado intervalo de tempo. Ao tratar de uma causa em particular (como a COVID-19), fala-se em uma Mortalidade causa-específica. Importante destacar que, para este indicador, leva-se em conta a população total (ou seja, tanto pessoas acometidas pela causa de morte em questão quanto as não afetadas). Desta maneira, a Mortalidade causa-específica ilustra o efeito de uma causa de morte no total da população estudada, fornecendo uma noção do risco à saúde coletiva. (para uma medida do efeito de uma causa de morte especificamente na população afetada, ver letalidade)
Mutações
Mutações são erros no processo de duplicação do material genético. Como o DNA (ou RNA) que compõe o genoma de um ente biológico (seja um vírus ou um ser vivo) é “copiado” por enzimas sujeitas a erros, pequenas mudanças podem ocorrer no processo, resultando em genomas ligeiramente diferentes do original. A mutação é um dos principais mecanismos de geração de diversidade na biologia, já que se as cópias fossem sempre exatas, não surgiriam diferenças entre organismos.
Essas diferenças geralmente são neutras ou prejudiciais, mas, caso eventualmente alguma alteração aumente o sucesso da próxima geração, a tendência é que essa mutação seja selecionada e torne-se mais prevalente com a passagem do tempo. No caso de um vírus, por exemplo, uma mutação que resulte em uma maior eficiência na entrada em células hospedeiras, ou na estabilidade do capsídeo viral — fazendo com que consigam se manter viáveis por mais tempo no meio celular — tende a tornar mais bem-sucedidas as variantes que a apresentam do que aquelas sem a mutação (para um esquema ilustrando este processo, ver Evolução do Genoma Viral acima). Mutações que levam a este tipo de diferença normalmente afetam diretamente a estrutura de algum gene, a unidade fundamental da informação genética.
Uma mutação pode também permitir que um vírus consiga entrar em outros tipos de células, e mesmo outras espécies, como no caso do SARS-CoV-2, que provavelmente se originou em morcegos e desenvolveu posteriormente a capacidade de causar doença e ser transmitido entre seres humanos. Outros exemplos são as gripes suína e aviária, que têm uma história similar.
Mutações definidoras de linhagem
Mutação ou conjunto de mutações que são características de uma linhagem, constatadas por sequenciamento genético nas amostras pertencentes a esta. Mutações definidoras de linhagem podem levar a genomas virais com características distintas, como uma maior ou menor transmissibilidade, de acordo com os genes afetados.
Pandemia
Apesar de o termo “Pandemia” possuir um significado lato sensu, uma revisão de 2009 voltada a entender os principais atributos que fazem com que um evento epidêmico seja de fato considerado uma “Pandemia” lista as seguintes características:
- “Ampla extensão geográfica” (evento sem fronteiras);
- “Mobilidade e Expansibilidade da Doença” (ou seja, a capacidade de ser competentemente transmitida por hospedeiros de uma localidade para outra);
- “Taxas elevadas de ataque e transmissão em surtos/irrupções” (múltiplos casos aparecendo em um curto intervalo de tempo);
- “Imunidade reduzida (ou ausente) na população”;
- “Originalidade e Evasividade do agente etiológico” (não necessariamente emergência de uma nova espécie, podendo ser uma evolução, uma nova linhagens ou sorotipo, ou simplesmente o recrudescimento de um mesmo patógeno após um certo período de tempo em que a população deixa de ser imunologicamente competente);
- “Infecciosidade” (a capacidade de causar uma infecção e se estabelecer);
- “Contagiosidade” (a capacidade de ser transmitida diretamente de uma pessoa a outra, com a ressalva de que pandemias como a da peste bubônica durante a Idade Média teve como principal rota de transmissão a via vetorial);
- “Gravidade dos sintomas e Letalidade” (com elevado impacto na capacidade produtiva da população e significantes taxas de óbitos);
É mais do que plausível atribuir-se todas as características acima de “Pandemia” a esta causada pela emergência do novo coronavírus humano, chamado de SARS-CoV-2 e que causa a Doença do Coronavírus 2019, simplesmente denominada de COVID-19. A COVID-19 é um agravo de grande preocupação, que ocasiona frequentemente quadros inflamatórios graves e óbitos. Sua provável origem é zoonótica, e sua alta adaptabilidade em infeccionar o hospedeiro humano, ser transmitido por via aérea, combinado ao fato de se tratar de uma nova espécie de vírus (originalidade) para o qual não nunca houve exposição prévia e consequentemente nenhuma imunidade adquirida (população imunologicamente virgem — também chamada pelo termo técnico naïve), contribuiu para a sua rápida disseminação por todos os continentes, facilitado também pela ampla mobilidade das populações hospedeiras em função de uma economia globalizada e um baixo nível de preparo e resposta (preparedness & response) dos sistemas de vigilância da grande maioria dos países, que não tinham maneiras de antecipar o novo patógeno, nem foram capazes de agir rapidamente o bastante.
Em termos técnicos, se posteriormente um agente causador de uma Pandemia (ou Epidemia) estabelecer-se localmente em uma dada população em aparente perenidade, eventualmente causando ciclos de infecções, sejam eles sazonais ou esporádicos, sem erradicação total do agente etiológico, denomina-se então a este padrão de “Endemia”.
Partículas de vírus infecciosas
Partículas virais capazes de se multiplicar em uma célula e/ou tecido, gerando um processo infeccioso, com a estrutura integral, incluindo proteínas do capsídeo, envelope fosfolipídico (quando presente), proteínas ou glicoproteínas de superfície — envolvidas no processo de invasão das células hospedeiras — e o genoma viral completo.
Em teoria, uma única partícula viral viável é capaz de iniciar o processo de infecção, ao encontrar uma célula hospedeira compatível, embora seja mais comum que diversas partículas adentrando o organismo de um hospedeiro sejam necessárias para que o processo de infecção tenha sucesso.
Partículas inviáveis
Partículas virais que, devido à ausência de um ou mais de seus constituintes, são incapazes de completar o ciclo viral com sucesso. Essa incapacidade pode se dar, por exemplo, pela produção de capsídeos vazios (sem o material genético), ou pela ausência de uma glicoproteína de superfície importante para o processo de invasão de células hospedeiras, ou ainda por moléculas defeituosas (tanto com material genético incompleto ou inviável quanto com proteínas ou glicoproteínas com a função comprometida por mutações de resultado deletério).
Patógeno Endêmico / Infecção Endêmica
Uma doença (ou o patógeno causador desta doença) pode ser considerado endêmico em uma localidade quando se mantém nela regularmente, com baixa prevalência. Eventualmente, uma doença endêmica pode causar surtos na região — ou seja, ter um aumento no número de casos — mas, ao contrário de surtos de doenças não-endêmicas, após a queda no número de casos a doença endêmica permanece circulando entre
reservatórios e hospedeiros, mantendo-se na baixa prevalência característica de uma endemia.
Preprint
Publicação inicial, contendo o texto e os dados similares aos de um artigo finalizado, mas publicada antes do processo de revisão pelos pares. Por não ter passado pelo rigoroso processo de verificação e análise independente por outros grupos de pesquisadores — parte do processo editorial regular para a publicação de artigos científicos — as conclusões de um pre-print são limitadas.
Este modelo de publicação é importante para o compartilhamento rápido de informação entre especialistas em meio a uma crise como a presente pandemia, e pode informar a mídia e a opinião pública desde que as informações sejam tratadas com cautela e responsabilidade extra, ressaltando o fato de que o processo editorial completo ainda é necessário para que se ganhe mais segurança nas conclusões apresentadas.
Prevalência
A prevalência de uma doença ou de uma linhagem viral é uma medida do número de pessoas afetadas pela doença em um determinado período de tempo. Uma linhagem é mais prevalente do que outra quando, em um dado momento, mais pessoas estão doentes por conta dela do que da outra.
Profundidade de Leitura
Uma medida da confiabilidade de uma sequência genética. A profundidade de leitura é a quantidade de vezes que um nucleotídeo foi detectado naquela posição, dentre todas as leituras de um processo de sequenciamento. Uma alta profundidade significa que a probabilidade de erro (ou seja, que um nucleotídeo numa dada posição não seja aquele apontado pelo sequenciador) é baixa.
R0
Também chamado de Número Básico de Reprodução, em epidemiologia o R0 define-se como a capacidade de espalhamento de um agente etiológico, assumindo que a população é 100% suscetível à doença. O número é uma estimativa de quantas pessoas, em média, se infectam ao entrar em contato com um paciente infectado. Portanto, um R0 de 1 significa que cada pessoa, ao longo do período de infecção, é capaz de transmitir a doença para uma única pessoa.
Quando o R0 está acima de 1, a tendência é que a doença se espalhe de forma a causar um surto (o número de casos aumenta, pois cada paciente infecta mais de uma pessoa), enquanto um R0 abaixo de 1 significa uma tendência de desaparecimento da doença.
Re
Enquanto o R0 é uma medida do potencial máximo de espalhamento de uma doença infecciosa, assumindo que não há nenhuma imunidade na população e que não há medidas de contenção do contágio, o Re é uma estimativa do número efetivo de contágios. O Re pode variar ao longo do tempo, de acordo com o desenvolvimento de imunidade (por vacinação ou em resposta à infecção), e a adoção de políticas públicas para contenção da doença e profilaxia — medidas que levam à prevenção da infecção.
No contexto da Covid-19, medidas preventivas, como o distanciamento social com adoção do trabalho não-presencial, a interrupção temporária de atividades escolares e, em situações mais extremas, o lockdown (situação em que todas as atividades presenciais não-essenciais são suspensas e a movimentação da população fica restrita a situações de extrema necessidade), são voltadas à redução do Re, ou seja, redução do número de pessoas efetivamente contaminadas pelo contato com um paciente.

Ao se tratar de doenças infectocontagiosas como a COVID-19, eventos nos quais muitas pessoas se aglomeram podem resultar em um aumento do Re, devido ao grande contato entre pessoas infectadas e outras susceptíveis. A eventos como este, dá-se o nome de “Evento de Supercontágio”.
Reservatório
Em epidemiologia, diz-se que um animal, uma pessoa ou mesmo um fator ambiental (ex.: um corpo d’água contaminado) é um reservatório para uma doença quando mantém o agente causador desta doença em circulação em longo prazo. O agente infeccioso pode ser transmitido, direta ou indiretamente, de reservatórios para hospedeiros da doença, podendo gerar surtos e até pandemias. Quando se trata de um reservatório biológico (pessoa ou animal), não é incomum que o reservatório em si não adoeça, servindo apenas como uma “ponte” para transmissão da doença a hospedeiros susceptíveis. Reservatórios também são um importante fator que dificulta a erradicação de uma doença, porque, mesmo que o patógeno seja completamente eliminado entre os hospedeiros da doença, ele permanecerá circulando entre reservatórios.
RT-PCR
Exame utilizado para detectar a presença e a quantidade de RNA em amostras biológicas. Através do uso de moléculas de RNA sintetizadas em laboratório, chamadas de “sondas” e “iniciadores”, que se ligam especificamente a uma sequência genética de interesse — neste caso, trechos do genoma do SARS-CoV-2 — é possível detectar se existe RNA viral na amostra e quantificar o número de cópias da sequência de interesse através do uso de fluorescência.
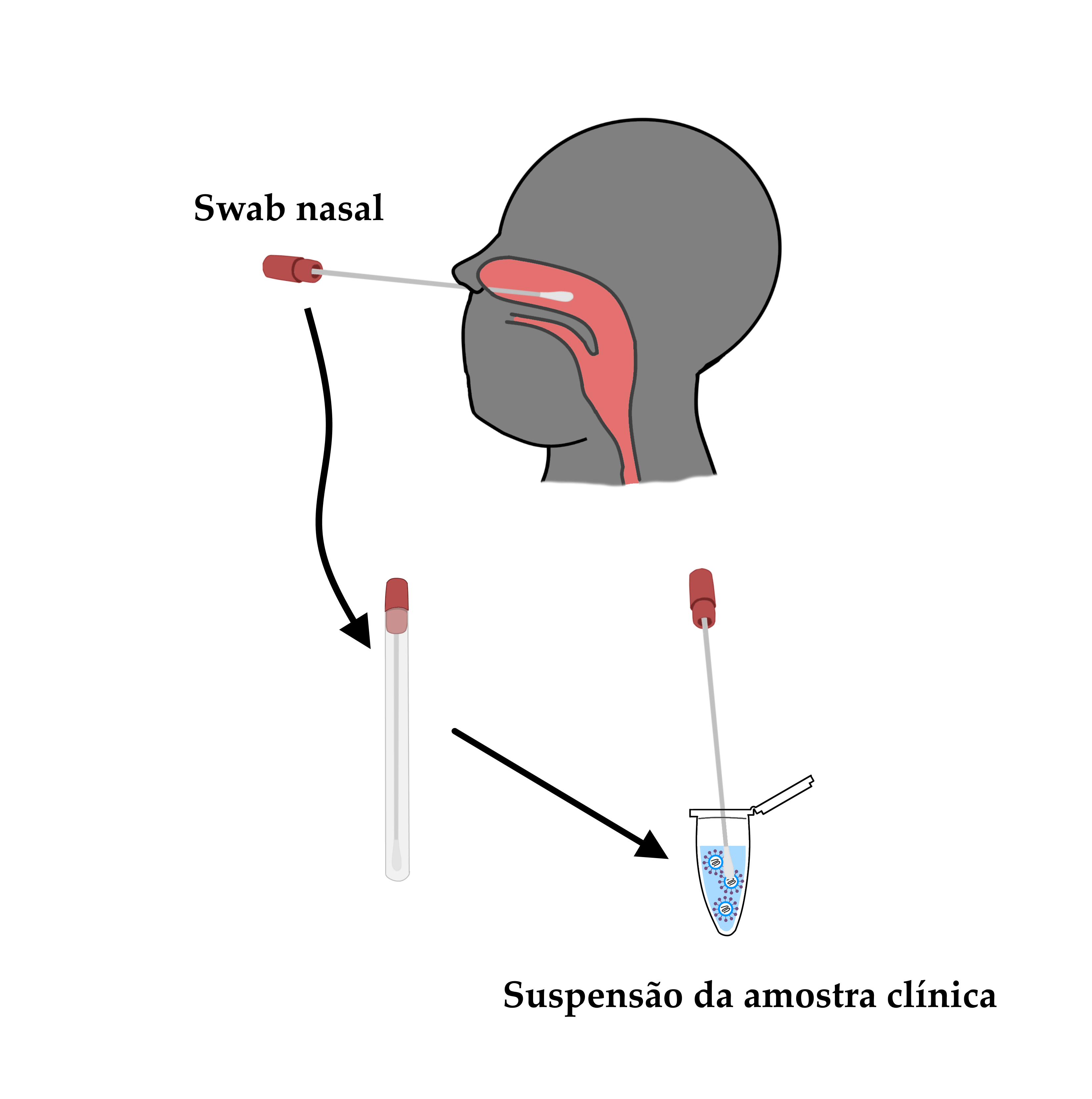
A obtenção de amostra clínica para detecção do material genético viral se dá pela técnica do swab nasal. Com o uso de um cotonete apropriado, profissionais de saúde coletam material da mucosa da orofaringe do paciente em caso suspeito, a partir do qual se obtém uma suspensão de partículas virais.
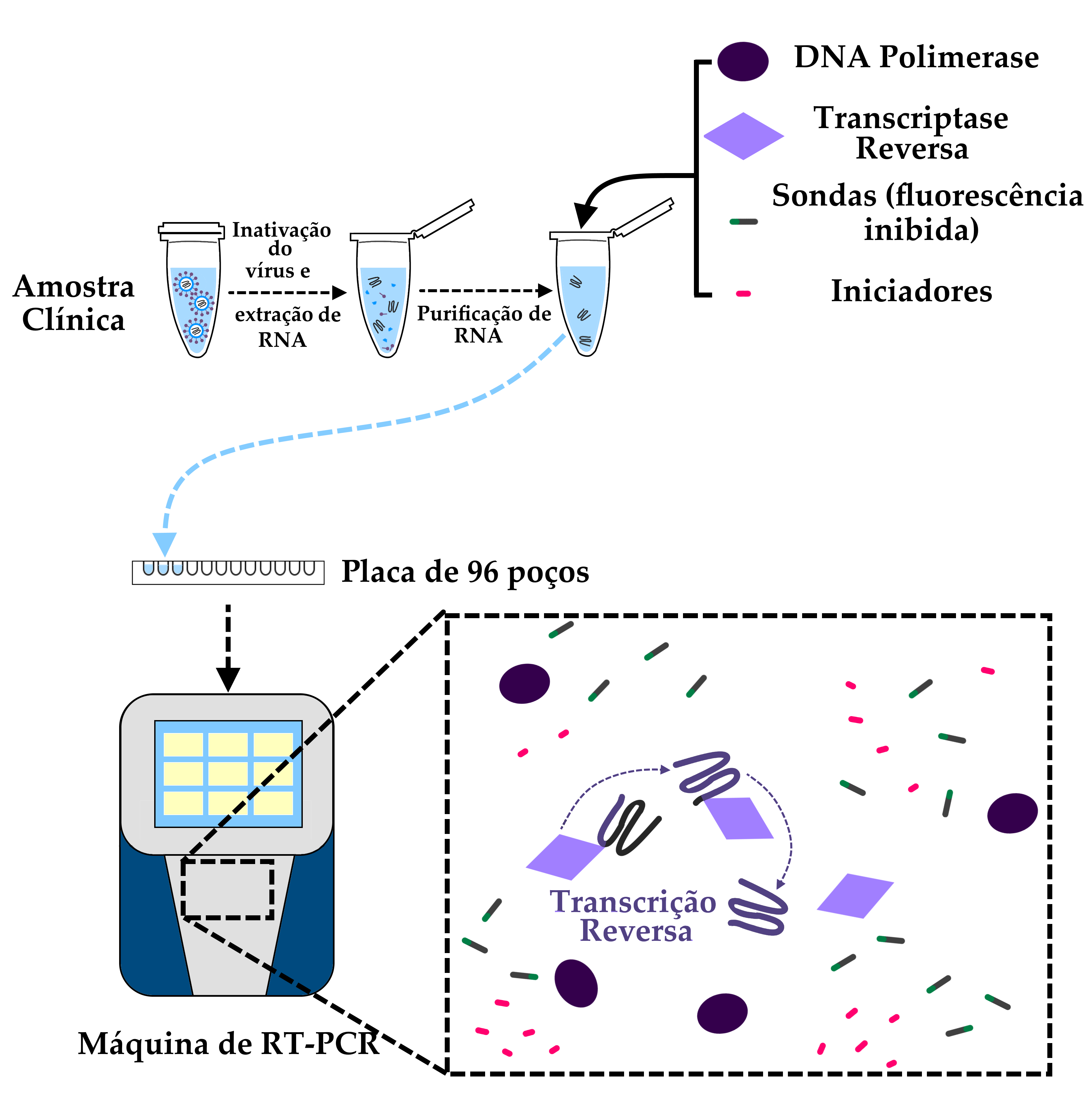
A partir da amostra clínica, é possível realizar a extração e purificação do RNA Viral. Com o RNA purificado, os reagentes necessários para a realização do RT-PCR são acrescentados ao “mix”, que é distribuído em uma placa de acrílico específica para este uso. Já no equipamento de RT-PCR, é obtido o chamado DNA complementar pela reação da Transcriptase Reversa (RT, que dá nome à técnica). É este DNA complementar — uma cópia fidedigna do material genético viral — que servirá como base para as etapas posteriores.
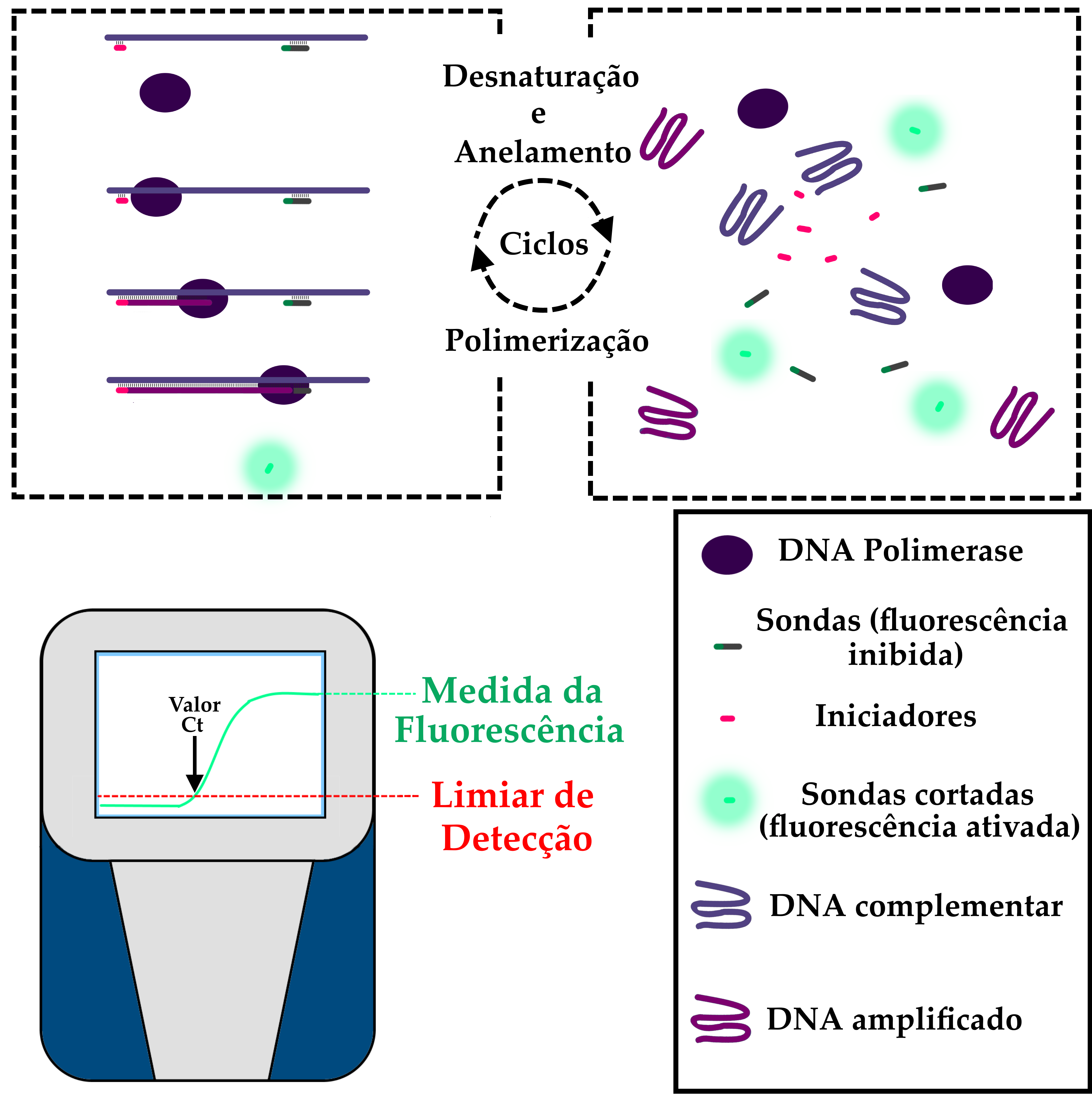
O aparelho de RT-PCR realiza diversos ciclos, durante os quais diminuições e aumentos controlados na temperatura das amostras permitem que as enzimas DNA-Polimerase façam cópias do DNA complementar. Ao copiar as sequências, a enzima promove a quebra das sondas, o que ativa os marcadores fluorescentes. Desta forma, a intensidade da fluorescência a cada ciclo é diretamente proporcional à quantidade de material genético amplificado. Quando a intensidade desta fluorescência torna-se suficiente para que os sensores do aparelho a detectem, diz-se que foi atingido o limiar de fluorescência. O número de ciclos necessários para que uma amostra atinja este limiar recebe o nome de “valor Ct”.
SARS-CoV-2
Do inglês, Severe Acute Respiratory Syndrome CoronaVirus 2, ou CoronaVírus da Síndrome Aguda Respiratória Grave 2. O causador da atual pandemia recebeu este nome por ser o segundo coronavírus responsável por uma epidemia caracterizada pela síndrome respiratória em seres humanos, após o surto epidêmico de 2003 na China, causado pelo SARS-CoV. A síndrome aguda respiratória grave é um quadro clínico que apresenta risco elevado à saúde e pode levar à morte.
Todas as linhagens e variantes do novo coronavírus pertencem à espécie SARS-CoV-2.
Semana epidemiológica
O calendário de semanas epidemiológicas consiste em um sistema de padronização da contagem de semanas, para facilitar a comparação entre diferentes anos no que diz respeito a estatísticas epidemiológicas (por exemplo, comparar a mortalidade por uma doença entre dois ou mais anos). Por convenção internacional, semanas epidemiológicas são contadas de domingo a sábado, sendo a primeira semana do ano a primeira a conter mais dias em janeiro e a última aquela a conter mais dias em dezembro.
Sequenciadores
Equipamentos que permitem desvendar a sequência de partes ou da totalidade de um genoma, a partir da leitura das sequências de nucleotídeos (“blocos” a partir dos quais o DNA ou RNA são construídos). Sequenciadores são baseados na reação de síntese do código genético, que ocorre de uma maneira muito similar quando células se dividem, aliada a tecnologias para identificar qual nucleotídeo está sendo inserido de cada vez. A obtenção de sequências de trechos do genoma, ou de seu conteúdo integral, permite comparar diferentes amostras, entender como funcionam as relações entre os genes e classificar amostras de acordo com suas relações de parentesco.
Sequências consenso
Também conhecidas como sequências canônicas, sequências consenso, na biologia molecular, são resultado da comparação de várias sequências de genoma alinhadas. Para formar uma sequência consenso, são considerados os nucleotídeos mais frequentes em cada posição. Assim, sequências consenso podem servir de referência para a análise de novas amostras.
Sistema de nomenclatura
Um conjunto de regras que devem ser seguidas para dar nome a uma variante recém-encontrada de um vírus. Para o SARS-CoV-2, a Rede Genômica Fiocruz segue o sistema de nomenclatura PANGO, proposto pelo Centro para Vigilância Genômica de Patógenos (Inglaterra).
Soroconversão
Também conhecida como “soroconversão imune”, consiste no momento em que anticorpos específicos — produzidos em resposta à exposição natural ou à vacinação contra um agente infeccioso ou toxina — podem ser detectados no plasma sanguíneo de um paciente. Em geral, a produção de anticorpos, feita por células chamadas linfócitos B, começa após um intervalo de tempo de alguns dias após a exposição. No primeiro momento, o mais comum é que sejam produzidos anticorpos do tipo IgM e, mais tardiamente, anticorpos do tipo IgG, de forma a ser possível distinguir entre eventos recentes de soroconversão (presença de IgM e pouco ou nenhum IgG) e eventos mais antigos (caracterizados por presença de quantidades maiores de IgG, e IgM em pequenas concentrações, ou mesmo indetectável).
SNP
Polimorfismo de um único nucleotídeo (do inglês Single Nucleotide Polymorphism) é a substituição de um único nucleotídeo em um gene, quando se comparam duas ou mais amostras. Se, por exemplo, em uma população, na posição X de um gene pode haver uma Timina (T) ou uma Adenina (A), pode-se dizer que há um SNP nesta posição do gene. Essas diferenças podem influenciar no sucesso evolutivo de vírus através de: alterações em proteínas que confiram vantagens ou desvantagens adaptativas — por exemplo, as diferentes alterações da proteína S que permitem que algumas variantes do SARS-CoV-2 causem reinfecção; ou uma fuga de mecanismos de defesa das células hospedeiras contra material genético invasor (como RNAs de interferência).
Taxa de Transmissão
Em modelos epidemiológicos, a taxa de transmissão é a proporção de indivíduos de uma população classificados como “suscetíveis” que passam para o estado de “infectados”. A taxa de transmissão, portanto, representa o espalhamento de uma epidemia através da população, e é diferente do R0 e do Re, embora esteja diretamente relacionada a ambos — por exemplo, medidas de distanciamento e a vacinação, pela redução dos valores de Re, também reduzem a taxa de transmissão. Outro exemplo é que patógenos de R0 mais alto levam a taxas de transmissão mais altas, dado que as outras condições (como a intensidade e adesão a medidas de isolamento e a suscetibilidade da população) sejam as mesmas.
Tempestade de Citocinas
Nome dado a um quadro clínico caracterizado por uma hiperativação do sistema imunológico, normalmente causada por uma alta produção de moléculas sinalizadoras de inflamação por parte de macrófagos alveolares. Estas moléculas sinalizadoras, denominadas “citocinas proinflamatórias” têm por função o recrutamento de mais células imunes que, por sua vez, podem produzir ainda mais citocinas, e iniciar processos inflamatórios que podem levar a danos generalizados pelo corpo, febre alta, falência de órgãos e óbito.
A tempestade de citocinas pode apresentar-se em doenças causadas por diversos patógenos — como nos quadros graves de dengue e de COVID-19, a peste bubônica e infecções bacterianas generalizadas —, doenças autoimunes e outros distúrbios do sistema imunológico, ou mesmo em resposta a terapias como o transplante de linfócitos-T para terapia antitumoral contra alguns cânceres.
O manejo de casos de hiperativação do sistema imunológico pode ser feito com o uso de imunomoduladores, e requer hospitalização e cuidados intensivos para manter pacientes com vida.
Valor Ct
Uma reação de PCR (seja o quantitativo em tempo real ou o PCR convencional), o equipamento realiza ciclos de mudança de temperatura, pois cada etapa da amplificação de material genético é otimizada em uma temperatura diferente. Em um RT-PCR, é possível acompanhar quantos ciclos se passam até que se tenha material genético o bastante para detecção pelo aparelho. Como uma maior quantidade de RNA numa amostra faz com que menos ciclos sejam necessários para se atingir esse limite mínimo de detecção, o número de ciclos, ao qual se dá o nome de Valor Ct (do inglês cycle threshold, em tradução livre “limite mínimo de ciclos”).
Desta maneira, como o Valor Ct é inversamente proporcional à quantidade de material genético analisado, no âmbito do monitoramento do SARS-CoV-2, podem-se comparar diferentes amostras quanto ao seu Valor Ct para avaliar quais têm uma maior carga viral (menor valor Ct). É importante ter em mente que esta não é uma medida direta do número de cópias do vírus no organismo de pacientes, mas um indicativo consistente que permite análises comparativas.
Variantes genéticas
Qualquer vírus que foi sequenciado e possui mutações que o diferenciam da versão original do vírus. O termo variante não implica importância epidemiológica e pode ser utilizado de forma generalizada para se referir a diversidade genética de uma espécie viral, ou seja, o repertório de diferentes versões de seus genes. O termo cepa também é utilizado pela mídia para denominar variantes virais, embora o uso deste termo não seja um consenso na comunidade virológica.
Uma lista, baseada na classificação atual da OMS das Variantes de maior relevância mundial para a pandemia COVID-19, está disponível em nosso site na página sobre as linhagens
Vírus
Grupo de entidades biológicas não-celulares. Não possuindo um metabolismo próprio (ou seja, sem organelas e enzimas para realizar reações químicas, gerar ou consumir energia), os vírus são parasitas, completamente dependentes de células hospedeiras para sua replicação — o processo de criação de novas cópias, análogo à reprodução. Desta forma, todo vírus consiste em um material genético envolto por uma estrutura de proteínas chamada de capsídeo.
Alguns vírus — como o causador da COVID-19, o SARS-CoV-2 — possuem uma camada de fosfolipídios chamada de “envelope”. O envelope é parecido com o envoltório celular, uma vez que porções da membrana da célula hospedeira são cooptadas por estes vírus envelopados para formar esta estrutura, ao final do processo de replicação. Com ou sem envelope, todo vírus precisa ter em sua superfície moléculas — geralmente proteínas ou glicoproteínas — utilizadas para se “agarrar” às células hospedeiras e colocar dentro delas o material genético viral, que pode ser baseado em DNA ou RNA, e contém toda a informação necessária para causar a infecção (incluindo genes para a produção das enzimas e proteínas virais, através das quais os parasitas dominam o metabolismo das células hospedeiras e o redirecionam para a produção de novos vírus)
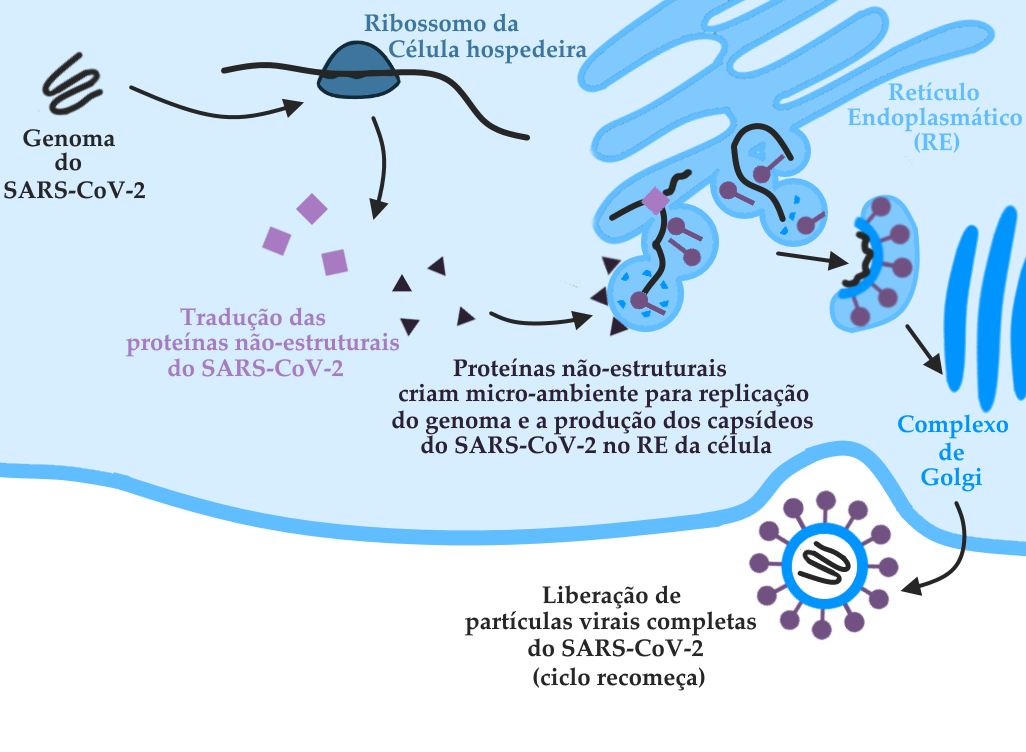
Esquema ilustrando as etapas da infecção posteriores à entrada do genoma viral na célula hospedeira. Atente para o fato de que o envelope viral é fruto da mistura entre proteínas do vírus e lipídeos de membrana da célula hospedeira.
VOC
Variantes de Preocupação (do inglês, Variants of Concern) são uma classificação dada a variantes dentro de uma linhagem, quando é demonstrada sua maior transmissibilidade (ou efeitos negativos para a epidemiologia do vírus), virulência (incluindo mudanças na apresentação clínica da doença) ou uma diminuição da efetividade de medidas de saúde pública (incluindo distanciamento social e as possibilidades diagnósticas, vacinais e terapêuticas disponíveis no momento) frente à variante em questão. Desta forma, variantes como a Gama (P.1), a Alfa (B.1.1.7) e a Beta (B.1.351), devido a seu espalhamento rápido em seus países de origem, ao alto número de mutações na proteína S e ao crescente número de evidências de sua capacidade de escape de anticorpos, foram classificadas como VOCs.
VOI
Variantes de Interesse (VOI, do inglês Variants of Interest) são uma classificação dada a perfis genéticos mutantes com potencial de gerarem uma piora no quadro epidêmico, mas que ainda não tiveram este potencial demonstradamente concretizado. A linha de corte de classificação é baixa — ou seja, bastam poucas mutações em genes relevantes bastam para que uma amostra receba esta classificação — para que variantes com a possibilidade de causar agravamento da questão sanitária não passem desapercebidas. Para ser classificada como VOI, uma variante tem de ter uma mutação que gere alterações de aminoácidos possivelmente associadas a mudanças na transmissibilidade, virulência (capacidade de gerar sintomas graves), epidemiologia, ou na capacidade de ser reconhecida pelo sistema imunológico, além de ter causado múltiplos casos da doença / transmissão comunitária.
VUM
Variantes sob Monitoramento (VUM, do inglês Variants under Monitoring) são uma classificação dada a perfis genéticos mutantes do SARS-CoV-2 que não necessariamente apresentam risco, mas têm características que justificam um monitoramento mais cauteloso. A Organização Mundial da Saúde classifica linhagens como VUMs quando suspeita-se que algumas de suas alterações genéticas tenham efeitos em características virais com potencial risco no futuro, mas sem evidências conclusivas sobre o risco aumentado associado a essas mutações, baseado no conhecimento atual.